通讯作者:蒋晓飞,联系电话:021-52888316。
作者简介:仉 英,女,1989年生,硕士,主要从事微生物致病机制研究。
建立敲除临床肺炎克雷伯菌耐药菌株质粒上的耐药基因的方法。
方法聚合酶链反应(PCR)分别扩增试验菌株待敲除目的基因
不同多位点序列分析(MLST)型别的2株临床肺炎克雷伯菌耐药菌株的
λ red同源重组法可以可靠敲除临床肺炎克雷伯菌耐药菌株质粒上的基因。
To establish a method of knocking out drug-resistant genes on plasmid contained by clinical
Polymerase chain reaction(PCR) was used to amplify the upstream and downstream fragments of target gene
The
Lambda red homologous recombination method can be used to knock out clinical
由于抗菌药物的滥用,导致临床菌株的耐药性不断增强。最主要的耐药机制是通过质粒或其它可移动原件获取并不断积累各种耐药基因,导致同一菌株携带多种耐药基因而产生多重耐药、泛耐药。我们研究小组通过研究2006年8月至2009年12月共57株泛耐药临床肺炎克雷伯菌株,发现其主要为2种多位点序列分析(multilocus sequence typing, MLST)型别:ST423型(HS062105)和ST11型(HS082416),所有菌株都携带KPC-2、CTXM-14和SHV-12型β-内酰胺酶基因。为研究泛耐药临床肺炎克雷伯菌积累多种β-内酰胺酶基因的原因,以及每种耐药酶在耐药机制中所起的作用和其相互之间的作用,将这些基因从临床菌株中逐一敲出,并逐一回补到肺炎克雷伯菌标准菌株中,这种快速、高效的研究方法目前尚未见报道。本研究小组成功地运用λ red 同源重组法敲除了一株ST423型(HS062105)和一株ST11型(HS082416)肺炎克雷伯菌临床菌株质粒上的 KPC基因。
肺炎克雷伯菌HS062105(ST423型)来自华山医院2006年8月从尿标本中分离到的临床菌株,肺炎克雷伯菌HS082416(ST11型)于2009年10月从痰标本中分离。质粒pKOBEG-Apr、pMQ300来自于英国Leicester大学实验室。
Bio-tech 公司生产的聚合酶链反应(polymerase chain reaction,PCR)仪、电导入仪、离心机,抗菌药物氨苄西林(ampicillin,AMP)、头孢噻肟(cefotaxime,CTX)、头孢他啶(ceftazidime,CAZ)、亚胺培南(imipenem,IMP)、美罗培南(meropenem,MEM)和厄他培南(ertapenem,ETM)购自中国药品生物制品检定所,血琼脂平板购自上海科玛嘉生物有限公司,菌株的分离与鉴定使用法国生物梅里埃公司全自动微生物分析系统VITEK-Compect,PCR试剂购自大连宝生物TaKaRa公司。引物合成由上海英骏生物有限公司完成,电泳结果分析使用Tanon2500数码凝胶图像分析系统。
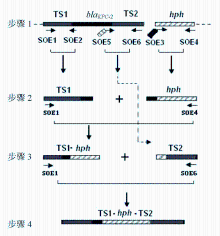
以试验临床菌株质粒为模板,SOE1、SOE2和SOE5、SOE6为引物,质粒pMQ300为模板,SOE3、SOE4为引物,分别进行PCR扩增,见表1,得到PCR产物TS1、TS2和 hph,然后以PCR产物TS1及 hph为模板,SOE1、SOE4为引物进行重叠延伸基因扩增(gene splicing by overlap extension polymerase chain reaction, SOE-PCR), 得到产物TS1- hph,再以TS1- hph为模板,以SOE1、SOE6为引物进行SOE-PCR即可得到产物TS1- hph- TS2。经鉴定的PCR产物用琼脂糖凝胶回收试剂盒进行回收纯化并测定DNA浓度,试验过程见图1[ 1, 2, 3]。
| 表1 试验引物 |
1. λ red依托质粒pKOBEG-Apr电转化进入试验菌株 试验菌株制备成为感受态,取0.1 μg pKOBEG-APr质粒DNA电击转化感受态细胞,电转后加入1 mL LB培养基,37 ℃振荡培养60 min之后涂于含阿普霉素的LB平板上,30 ℃过夜。30 ℃过夜LB平板上生长的单克隆即为含有λ red依托质粒pKOBEG-Apr的试验菌株。
2. λ red 重组蛋白的诱导 挑取30 ℃过夜LB平板上的单克隆,加入5 mL LB培养液中过夜培养,次日以1∶50的比例接种细菌至100 mL LB培养液中,30 ℃振荡培养至吸光度( A)600 值为0.2时,加入L-阿拉伯多糖至终浓度为0.1%,继续30 ℃振荡培养至 A600值为0.6,在此期间exo/bet/gam蛋白在L-阿拉伯多糖的诱导下发生了短暂表达,将诱导表达后的细菌制备成为感受态。
取0.2 μg带有同源臂的融合片段(步骤3)电击转化感受态细胞(步骤5),电转后加入1 mL LB培养基,30 ℃振荡培养60 min,最后取其中的100 μL涂于潮霉素(150 μg/mL)+阿普霉素(10 μg/mL)LB平板上,30 ℃过夜。30 ℃过夜LB平板上生长的单克隆同时含有λ red依托质粒pKOBEG-Apr和带有同源臂的融合PCR产物,在λ red 重组蛋白的诱导下,同源重组敲除试验菌株的 bla KPC-2 基因。
挑取数个单克隆至2 mL LB培养基中[潮霉素(150 μg/mL)+阿普霉素(10 μg/mL)],37 ℃振荡培养过夜,次日提取质粒 DNA,PCR扩增 bla KPC-2及 hph基因。
抽提原试验菌株及重组后菌株RNA,逆转录PCR定量检测 bla KPC-2基因的表达量。
参照美国临床实验室标准化协会(Clinical and Laboratory Standards Institute, CLSI) M07-A8,采用微量肉汤最低抑菌浓度(minimal inhibitory concentration,MIC)法检测野生株及 bla KPC-2缺陷突变株对AMP、CTX、CAZ、IMP、MEM和ETM的敏感性。
耐药基因片段、上下游片段及融合片段电泳结果见图2,泳道1~5为ST423型试验菌株各片段电泳图,泳道6~10为ST11型试验菌株各片段电泳图。
野生株中 bla KPC-2 基因为阳性, hph 基因为阴性,而 bla KPC-2缺陷突变株中 bla KPC-2 基因为阴性, hph 基因为阳性,即可确认 bla KPC-2缺陷突变株发生了同源重组,成功敲除了 bla KPC-2 基因,见图3。
使用逆转录PCR,以HS08763作为参考菌株,野生株HS062105(ST423型) blaKPC-2基因表达量为参考菌株表达量的1.9倍,野生株HS082416(ST11型) blaKPC-2基因表达量为参考菌株表达量的0.9倍。 blaKPC-2缺陷突变株ΔHS062105、ΔHS082416 blaKPC-2基因表达量检测不出,说明成功敲除了野生株 blaKPC-2 基因。
野生株HS062105(ST423型)、HS082416(ST11型)对AMP、CTX、CAZ、 IMP、MEM、ETM均耐药, bla KPC-2缺陷突变株ΔHS062105对IMP、MEM敏感,ΔHS082416对IMP、MEM、ETM敏感。见表2。
| 表2 野生株及 bla KPC-2缺陷突变株药物敏感性试验结果 |
传统的同源重组敲除某特定基因需要多步亚克隆,费时、费力。λ red重组质粒pKOBEG-Apr携带λ red,在L-阿拉伯糖的诱导下可表达同源重组蛋白exo/bet/gam,介导电转入的线性融合产物与相应的基因发生同源重组,然后再用合适的抗菌药物筛选,即可得到重组突变株,省时、省力。由于pKOBEG有一温度敏感的复制起始位点,当温度高于37 ℃时,细菌将消除质粒pKOBEG。不会对细菌后续研究造成干扰,是一种简单、高效的敲除方法。
泛耐药菌株对几乎所有临床常用的抗菌药物都耐药,很难找到合适的耐药标记筛选特定的突变克隆株,造成临床菌株的研究困难,进展缓慢。潮霉素和阿普霉素广泛应用于农业细菌学研究,未在临床中使用。临床菌株普遍对这2种药物敏感,因此在临床菌株中非常有用。
本研究选取了2006年8月至2009年12月分离的肺炎克雷伯菌HS062105(ST423型)、HS082416(ST11型),在这个时间段内,泛耐药肺炎克雷伯菌快速播散,临床分离菌株数量呈指数增长,并由ST423型转变为毒力更高的ST11型。研究耐药基因水平传播的机制,此时间节点分离到的泛耐药肺炎克雷伯菌最具代表性。我们成功地用FKPCU-Hyg-FKPCD片段替换了细菌的 KPC基因,克隆证实突变菌株KPC(-)、Hyg(+),药物敏感性试验结果显示菌株敲除 KPC基因后对IMP、MEM和ETM敏感,而菌株HS062105(ST423型)仅对IMP和MEM敏感,ETM仍耐药。究其原因是HS062105(ST423型)、HS082416(ST11型)还产生CTXM-14及SHV-12,因此对CAZ、CTX等仍耐药,HS062105(ST423型)对ETM仍耐药是因为该菌株还产生一种头孢菌素酶,有文献报道高表达头孢菌素酶加上膜孔蛋白缺陷可导致菌株对ETM耐药[ 4]。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|