通讯作者:应春妹,联系电话:021-68383297。
作者简介:姚冬婷,女,1989年生,学士,主要从事病原菌耐药机制研究。
评估微卫星多态性分析技术在临床光滑念珠菌基因分型中的作用。
方法收集2011年1月至2012年12月仁济医院和东方医院临床分离的光滑念珠菌59株,采用多位点序列分析(MLST)和微卫星多态性分析技术进行基因分型,比较2种基因分型方法的结果和分辨力。
结果59株光滑念珠菌经MLST分型得到6个序列型,其中ST-7型43株、ST-10型7株、ST-15型和ST-55型各3株、ST-3型2株、ST-43型1株,鉴别力指数(DP)为0.456;经微卫星多态性分析方法分为10型,其中A型25株、B型10株、C型8株、D型6株、E型3株、F型和G型各2株、H~J型各1株,DP为0.770。
结论微卫星多态性分析方法简便、快速,分辨力高于MLST技术,可作为实验室基因分型的首选方法。
To evaluate the application significance of microsatellite polymorphism in genotyping of
From January 2011 to December 2012, 59 isolates of
By MLST, the 59 isolates belonged to 6 clone sequences, including 43 isolates with ST-7, 7 isolates with ST-10, 3 isolates with ST-15, 3 isolates with ST-55, 2 isolates with ST-3 and 1 isolate with ST-43, and the index of discriminatory power (DP) was 0.456. By microsatellite polymorphism, the 59 isolates were classified into 10 genotypes. There were 25 isolates with type A, 10 isolates with type B, 8 isolates with type C, 6 isolates with type D, 3 isolates with type E, 2 isolates with type F, 2 isolates with type G, 1 isolate with type H, 1 isolate with type I and 1 isolate with type J, and the DP was 0.770.
ConclusionsMicrosatellite polymorphism is a simple and rapid method for molecular typing with higher discrimination than MLST. Therefore, microsatellite polymorphism can be the preferred choice in clinical laboratories.
光滑念珠菌是一种人体黏膜组织的非致病性共生菌,只是偶尔引起机会感染。近年来,由于免疫疗法和广谱抗真菌药物的广泛使用,光滑念珠菌已经成为一个重要的条件致病菌,快速、准确的分型技术对预防监测流行病学调查具有重要意义。目前已有多种分子生物学基因分型技术,包括微卫星多态性分析(microsatellite polymor-phism)、脉冲场凝胶电泳(pulsed field gel electro-phoresis)、限制性片段长度多态性(restriction fragment length polymorphism, RFLP)、多位点序列分析(multilocus sequence typing, MLST)等。本次研究通过比较微卫星多态性分析技术和MLST技术在光滑念珠菌基因分型中的应用,旨在为临床提供更简单、有效的基因分型方法。
收集2011年1月至2012年12月在仁济医院和东方医院临床分离的光滑念珠菌59株,其中痰标本25株,中段尿8株,血液7株,分泌物6株,咽拭和粪便各4株,肺泡灌洗液、引流液、颈静脉导管、棉拭和清创物各1株。所有菌株均经API板条(法国生物梅里埃公司)鉴定为光滑念珠菌。
1. DNA提取 从沙保弱培养基中挑取3~5个菌落转入300 μL生理盐水中混匀,2 000× g离心3 min,收集沉淀。按照真菌DNA基因组提取试剂盒(Biospin Fungus Genomic DNA Extraction Kit,上海Bior, Cat。No: BSC14S1)步骤进行操作,最后将抽提的DNA于-20 ℃保存备用。
2. MLST检测 采用Dodgson等[ 1]推荐的方法确定光滑念珠菌基因组上的6个管家基因( FKS、 LEU2、 NMT1、 TRP1、 UGP1、 URA3)为目的基因片段,见表1。聚合酶链反应(polymerase chain reaction,PCR)采用25 μL体系,其中10×PCR 缓冲液(含Mg2+)2.5 μL,2.5 μmol/L dNTPs 2 μL,10 μmol/L上游引物和下游引物各1 μL,TaKaRa Taq酶(5 U/μL)0.2 μL,模板DNA 1 μL,无菌蒸馏水加至25 μL。反应条件:94 ℃预变性7 min;94 ℃变性1 min,退火时间1 min(退火温度见表1),74 ℃延伸1 min,共30个循环;74 ℃总延伸10 min。
| 表1 MLST PCR引物序列及退火温度 |
将PCR扩增产物用1.5%琼脂糖凝胶进行电泳,EB染色后于凝胶成像仪上判读结果,观察是否扩增出目的条带。扩增出的目的核酸片段由华大基因进行正反向测序。
3. 微卫星多态性分析 采用Foulet等[ 2]推荐的方法确定光滑念珠菌基因组上的3个微卫星位点( RPM2、 MT1、 ERG3)作为目的基因片段,见表2。PCR采用20 μL体系,其中10×PCR缓冲液(含Mg2+)2 μL,2.5 μmol/L dNTPs 1.6 μL,10 μmol/L RPM2上游引物和下游引物各0.5 mol,10 μmol/L ERG3上游引物和下游引物各0.5 μL,10 μmol/L MT1上游引物和下游引物各2 μL,TaKaRa Taq酶(5 U/μL)0.25 μL,模板DNA 2 μL,无菌蒸馏水加至20 μL。反应条件:95 ℃预变性10 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,共30个循环;72 ℃总延伸5 min。扩增后的PCR产物送上海生工公司进行检测。
4. 鉴别力指数(index of discriminatory power, DP) 对于每种基因分型方法,计算其DP[ 3],以评估该方法将2株不相关菌株分到2个不同组别的能力。DP=1- 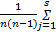
| 表2 微卫星序列 PCR荧光引物序列 |
59株光滑念珠菌均扩增出6个管家基因(FKS、LEU2、NMT1、TRP1、UGP1、URA3),各基因对应条带片段长度分别为589、512、607、419、616及602 bp。将每株菌的6个管家基因片段的测序结果与 MLST数据库进行比对,结果显示59株光滑念珠菌得到了6个序列型, ST-7型43株、 ST-10型7株、 ST-15型和 ST-55型各3株、 ST-3型2株、 ST-43型1株。见表3。
| 表3 MLST基因分型结果 |
光滑念珠菌是单倍体,因此在给定的3个位点(RPM2、MT1、ERG3)均只有1个峰值。等位基因分别命名为碱基对的长度。59株光滑念珠菌共分为10型, A型25株、 B型10株、 C型8株、 D型6株、 E型3株、 F型和 G型各2株、 H~J型各1株。见表4。
| 表4 微卫星多态性分析基因分型结果 |
在真核生物的基因组中,除了有编码蛋白质的单拷贝序列以外,还存在中度重复序列以及高度重复序列。高度重复序列中又存在由短的串联重复单位组成的序列,称为“串联重复序列”,其中串联重复序列又分为2种类型:小卫星 DNA和微卫星 DNA。小卫星 DNA是一些重复单位在11 ~60 bp、总长度从几百到几千个碱基组成的串联重复序列;微卫星 DNA是指由少数几个(1 ~5个)碱基对为重复单位,长几十个核苷酸的短串联重复 DNA序列,如( GA) n、( AT) n和( GAA) n等。微卫星多态性分析是根据特定微卫星基因序列设计引物, PCR扩增该微卫星位点的核心序列和部分侧翼序列测定 PCR产物的长度。 PCR产物长度的不同是由核心序列重复数不同而产生,从而进行基因组 DNA之间的多态性分析。
微卫星多态性分析作为一种新型的基因分型方法,已经广泛应用于光滑念珠菌的基因分型。 Foulet等[ 2]采用RPM2、MT1和ERG3这3个位点进行分型,将138株光滑念珠菌分为21型, DP达0 .84。 Abbes等[ 4]同样采用这3个位点将85株光滑念珠菌分为19型,在增加3个新的位点(GLM4、GLM5、GLM6)后,85株菌分为37型, DP达0 .941。 Grenouillet等[ 5]采用了另外6个新的微卫星位点( Cg4、 Cg6、 Cg10、 Cg5、 Cg7、 Cg1),127株光滑念珠菌分为37型, DP指数达0 .902。而 Brisse等[ 6]的报道采用了9个微卫星位点,将198株光滑念珠菌分为90个型, DP指数达0 .96。上述研究均提示微卫星多态性分析技术是一种有效的、分辨率高的基因分型方法。
MLST技术是指通过测序技术揭示管家基因的等位基因突变,从而达到对病原菌分型和鉴定的目的,便于不同实验室结果的相互比较,使全球的分子流行病学数据标准化,是目前公认最为权威和客观的基因分型方法。 Lin等[ 7]将80株光滑念珠菌进行 MLST分型,发现50株菌分布于 ST-7型、 ST-3型、 ST-10型、 ST-15型和 ST-26型,其余24株为新的 ST型,其中以 ST-7型居多,占30 %。 Lott等[ 8]也将 MLST分型技术用于光滑念珠菌,结果美国多个地区血培养中分离到的230株光滑念珠菌以 ST-16型、 ST-19型和 ST-3型为主。因此,应用 MLST方法可实现不同实验室试验分析结果之间的比较,为全球范围内大规模流行病学调查奠定了基础。
本研究以 MLST技术为参考方法评估微卫星多态性分析方法的应用: MLST和微卫星多态性2种分型方法分别将60株光滑念珠菌分成了6个和10个型别, DP分别为0 .456和0 .770。43株 ST-7型光滑念珠菌经微卫星多态性分析方法分成了3个单独的型别,7株 ST-10型菌分成了2个单独的型,3株 ST-15型菌也分成了2个单独的型。2种方法分型结果一致性高。因此,我们认为微卫星多态性分析方法作为一种新型的基因分型方法,能对光滑念珠菌进行有效的分型。与 MLST方法相比较,具有快速、简便、分辨力高等优点,可作为实验室大量菌株分型的首选方法。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|

