利用全基因组芯片扫描技术对染色体核型检测结果异常的患者样本进行重复检测分析,验证并确认患者染色体的具体核型。
方法利用基因分型芯片技术对9例临床表型皆为智力落后合并多发畸形,且核型结果异常的样本进行检测分析,比较两种技术之间结果相符程度,通过芯片平台结果来验证染色体核型技术的准确性,同时分析其临床适用性。
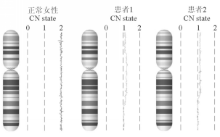
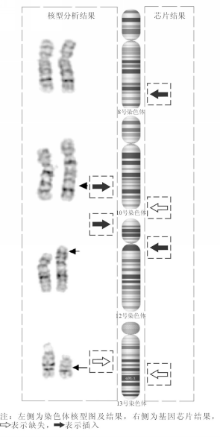
结果染色体核型结果和全基因组芯片分析技术的结果完全符合的为2例Turner综合征患者,均为嵌合型;3例染色体核型结果阳性患者,全基因组芯片分析结果为阴性,其中2例为随体增加,1例为染色体内倒位;4例涉及染色体片段大小不同的缺失和复制的患者,核型结果和全基因组芯片结果差异较大,并且核型检测结果与患者实际核型相差较大。
结论染色体核型技术在用于以往认定的适应症如智力落后、多发畸形的检测中,检测的准确性相对全基因组芯片技术较低,在明确定位染色体缺失和复制大小及位置的能力上有明显的不足,但对于检测染色体平衡性结构性变化的作用不能被芯片所取代。
To analyze the results of patients with chromosome abnormality by chromosomal microarray,and validate and depict patient's karyotype.
MethodsGenotyping array was applied to assess in 9 patients with hypophrenia and deformity. The samples with karyotype analysis abnormality were determined. Comparison between karyotyping and microarray analysis was considered to validate the clinical utility of chromosomal microarray analysis.
ResultsTwo Turner syndrome patients had completely concordant results from karyotype analysis and chromosomal microarray. Three patients showed abnormal karyotyping results (2 patients with excess trabant and 1 patients with chromosomal inversion) were detected negative with chromosomal microarray. Four patients with different size and location of deletions and duplication were also detected abnormal with chromosome microarray,but with significant deviation on location.
ConclusionsCompared with chromosomal microarray analysis,karyotype analysis has low accuracy in diagnosing patients with hypophrenia and deformity. The abilities of locating and reckoning chromosomal deletion and duplication by chromosomal microarray are insufficient for clinical diagnostic purpose,but karyotype analysis is still efficient in detecting balanced changes in chromosome.
一直以来,临床针对智力落后、多发畸形患者诊断的首选方法和金标准都是对培养的中期细胞核进行传统细胞遗传学的染色体核型分析。随着人们对细胞遗传学研究的深入了解,染色体核型技术的分辨率和准确性已经不能完全满足于临床诊断要求。近年来全基因组芯片技术(chromosomal microarray analysis,CMA)的发展和在临床的初步应用探索,展示了该技术平台无可比拟的优越性,全基因组芯片平台已经在产前和产后的诊断中展示出该平台在特异性和敏感性上的优势[
纳入本研究的样本共9例,其中男4例,女5例,年龄6月7岁,均为智力落后合并发育迟缓患者,部分患者同时有面部畸形的表现,如眼距宽、耳位低、通贯手和小头畸形等。所有患者均来自上海儿童医学中心遗传科或儿童保健科。
静脉采集受检患者外周血3 mL,肝素抗凝,无菌条件下将约0.3 mL抗凝血接种于RPMI-1640培养基,隔水式恒温培养箱37℃静置培养70 h,加入秋水仙素工作液0.05 mL(0.05 μg/mL)作用50 min后收获细胞,经低渗处理、固定作用后染色体标本制备,G显带技术进行核型分析。嵌合体计数100个中期分裂相。根据细胞遗传学国际命名体制(ISCN,1995年)进行识别和描述。
全基因组芯片试剂为Affymetrix CytoScan Assay Kit (Affymetrix,Santa Clara,CA)和Clontech TITANIUMTM DNA扩增试剂盒(Clontech,Mountain View,CA);芯片操作仪器包括聚合酶链反应(PCR)扩增仪GeneAmp PCR system 9700(Applied Biosystem,Fostercity,CA)、杂交炉GeneChip® Hybridization Oven 645,洗涤工作站GeneChip® Fluidics Station、 扫描仪GeneChip® Scanner 3000 7G(Affymetrix,Santa Clara ,CA)。具体的操作依照Affymetrix公司标准操作规程进行,包括样本限制性酶内切、引物结合片段连接、PCR扩增、PCR产物片段化处理、生物素荧光标记、杂交和洗涤等步骤。芯片结果分析软件为AGCC(Affymetrix GeneChip Convert Console)和CHAS(Chromosome Analysis Suite Version 2.0.0)。
9例患者异常核型结果2例为Turner综合征核型(均为嵌合型)45,X[
利用全基因组芯片技术检测该9例患者全基因组结构性变化结果:2例Turner综合征患者显示X染色体均为杂合性缺失,拷贝数(CN state)分别为1.07和1.08(图1);2例随体增大患者和1例染色体内倒位患者全基因组未见结构性变化;1例平衡易位患者染色体上出现多段缺失(chr1,1p31.1-31.3 ; chr4,4q32.3 ; chr11.11q14.1-14.3)。
3例微缺失或插入患者检测结果为:8号染色体插入合并10号染色体缺失(chr8:99804212-146295771 Gain 46.5mb 8q22.2-qter chr10:127441438-135427143 Loss 7.99mb 10q26.2-qter),12号染色体插入(chr12:29766862-50507802 Gain 20.7mb 12p11.22-q13.12 chr19:28271146-28829404 Gain 558kb 19q11-q12);13号染色体缺失(chr13:63187793-93266080 Loss 30mb 13q21.3-q31.3)(图2):
染色体核型技术一直以来是临床诊断先天性智力落后合并发育迟缓、以及多发畸形等相关适应症的首选方法,但是该方法的准确性和敏感度已无法完全满足临床诊断的需要。特别是基因芯片技术在平台发展和成本控制两方面做得越来越好的情况下,国外已经有大量临床诊断实验室将基因芯片替代染色体核型诊断技术,作为相关适应症的首选诊断方式。
本研究中的9例患者均以相同或相近的表型在本院遗传科或儿保科就诊,在染色体核型结果仅能提供比较模糊的诊断信息的情况下,我们采用基因分型全基因组芯片对这9例患者再次进行检测,通过全基因组芯片技术的检测结果,试图来验证染色体核型的结果,明确患者的遗传背景,帮助临床做出准确诊断。
9例患者中2种技术平台结果相符的只有2例,为Turner综合症患者,2例患者均为嵌合型X染色体缺失。值得注意的是,在嵌合程度的解释上,核型分析是通过计数不同核型的细胞,而全基因组芯片是通过相应染色体区域内探针拷贝数值的大小平均值进行预测。在大多数情况下,Turner综合征的患者可存在多种细胞核型嵌合的情况[
2例染色体核型分别报告为13号和15号随体增加的患者,全基因芯片分析结果为阴性,即全基因组未见明显的失衡现象。随体是识别染色体的主要特征之一,随体增加在人群中有多态性的存在,并不能直接作为患者致病的遗传背景作为临床诊断和解释临床症状。虽然有相关研究证明随体增大和耳聋或不良孕产史相关,但将随体增大作为对于临床智力落后,发育迟缓患者的诊断显然是不恰当的。另1例染色体内倒位的患者,全基因组分析结果也为阴性,提示在染色体断裂位点没有发生缺失,但断裂位点是否涉及具体的基因的情况,没有办法通过这2种技术平台检测到。最近发展起来的通过配对文库(mate-pair library)二代测序技术,可以同时检测到染色体平衡性和非平衡性的结构性变化[
另外4例患者染色体核型分析结果为微插入和微缺失,全基因组芯片分析结果与核型分析结果相差较大。其中1例患者染色体核型分析结果为10号染色体上有段缺失,但全基因组芯片结果为10号染色体插入,同时检测到染色体核型未检出的8号染色体上的大片段缺失;另2例患者全基因组芯片分析检测的微插入片段的大小和具体的染色体区带位置也与染色体核型分析结果存在较大偏差(图2),这可能是由于常用的G显带染色方法中秋水仙素处理的时间及浓度、低渗等对染色体显带的效果产生较大的影响,导致每次处理的效果都不相同[
通过9例患者染色体核型分析结果与全基因组芯片分析结果的比较,发现核型技术在诊断相关适应症的实际应用中存在准确性和敏感性不足的问题,而利用全基因组芯片分析技术,能对染色体非平衡性变化进行准确的检测和定位,对患者遗传背景的解释较为可靠,对临床诊断的帮助较大;但对平衡性染色体结构变化如倒位等,全基因组芯片无法检测到断裂点和涉及的基因。因此建议临床在诊断相关适应症时,可将染色体芯片技术作为首选技术,用于提高诊断效率,需要检测患者染色体平衡性变化时仍可采用染色体核型技术。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|