微小RNA(microRNA,miRNA)是一类长约20~24个核苷酸的小分子非编码RNA,其可以通过与特定mRNA结合或调节特定mRNA的蛋白质翻译过程来调控基因的表达[ 1]。越来越多的数据显示,miRNA在多种人类疾病中表达异常。然而,每种miRNA在不同环境中的特定功能还尚待发现。研究其功能的最好方法就是使关注的miRNA失去功能,“封闭海绵”(miRNA sponge)就是新近发现的具有封闭miRNA功能的物质。
1. miRNA的概念及特征 miRNA是一类由内源基因编码的长度为20~24 个核苷酸的非编码单链RNA 分子,其在体内参与转录后基因的表达调控。从发现至今已经有1 000余种miRNA在人类中鉴定出来[ 2]。miRNA首先由RNA聚合酶II转录成长达数百个或数千个碱基大小的初始转录子(pri-miRNAs)。初始转录子被含有Drosha(核酸内切酶III )和双链RNA结合蛋白Pasha的复合物剪切为60~70个的miRNA前体(pre-miRNA)[ 3]。miRNA前体由小分子单体G 蛋白Ran-GTP 依赖的转运蛋白exportin-5 运输至细胞质, 被Dicer酶(核酸内切酶)剪切成约22 个核苷酸的双链miRNA[ 4]。双链miRNA 被解旋酶解螺旋后,1条链被降解,另1条成为5′端为磷酸基团、3′端为羟基的成熟miRNA。
miRNA虽不编码蛋白,但通过对特定mRNA的降解或沉默来实现对特定基因的调控。计算证明,miRNA在一般情况下可以调控上百个基因。然而,每种miRNA在不同环境中的特定功能仍不清楚[ 5]。
2. “封闭海绵”的产生 研究miRNA功能的最好方法就是令待研究的miRNA失去功能。目前来讲有3种方法:基因敲除、寡核苷酸抑制剂、“封闭海绵”[ 6, 7, 8]。每种方法都各有优劣。寡核苷酸抑制剂主要用于短期实验;基因敲除虽可用于长期体内研究,但有相当数目miRNA位于蛋白编码区内或在miRNA簇中,所以基因敲除无论在时间、花费还是技术上都面临挑战;而包含多个微小RNA结合位点的“封闭海绵”作为一种可以替代基因敲除方法脱颖而出[ 9]。
“封闭海绵”含有多个高亲和力的miRNA的反义结合位点[ 23]。作为一个合成基因可以与miRNA的3′UTR区(Untranslated Regions,非翻译区)结合达到封闭特定miRNA功能。当“封闭海绵”在细胞内高表达时,它可以抑制有相同种子序列(miRNA中2~7个核苷酸决定着对靶基因识别的特异性)的miRNA家族活性。可以将编码miRNA基因彻底沉默[ 10],保证特定miRNA丧失活性。
第一个内源性的“封闭海绵”是在植物体内发现的,其可受环境因素介导miRNA表达的强弱[ 11]。2007年,Margaret S Ebert等[ 12]将含有miRNA结合位点的序列插入到由巨细胞病毒启动的编码报告基因GFP3′UTR区,制备出第一个人工合成的“封闭海绵”,为miRNA研究开辟了1条新途径。这段序列在第9~12位上有个凸出部位能阻止RNA干扰和“封闭海绵”降解,其也可以与miRNA核心区完美结合达到对miRNA功能的完全封闭。具有4个核苷酸组成的中央凸起的海绵比拥有完美序列反义结合位点的海绵之所以在封闭功能上更有效是因为后者会被内源性AGO2的裂解活动影响以至于产生海绵体的降解从而减弱封闭效果[ 24]。见图1、图2:
注:在miRNA海绵的第9~12位点处的凸起部位为错配位点。该错配位点位于RISC的切割位点上,从而阻断了海绵的降解.
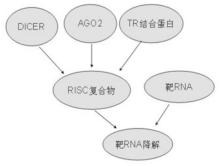
注:RNA诱导沉默复合物(RISC)由3部分组成:切割酶(Dicer)、Argonaute酶以及TR结合蛋白。当信使RNA与RISC蛋白复合体结合后,RISC开始切割信使RNA或是阻断信使RNA的翻译.
miRNA“封闭海绵”封闭效率不仅依赖结合位点间的亲和力还依靠海绵与miRNA间比例关系。为了使封闭效果最大化,应该使用所属细胞类型最强的促进剂,比如哺乳细胞中巨细胞启动子[ 12]。“封闭海绵”中凸起部位与促进剂合理选择构建出经典的miRNA海绵。
经典“封闭海绵”包括4~10个不相邻结合位点,位于RNA不编码的非结构区,之间由不等量核苷酸分开。增加位点的数目可以减少边际效应,但伴随结合位点增加,也同时增加封闭RNA降解几率[ 10]。在“封闭海绵”上人为制造突变可以导致一些部位错配,从而减少复制期间重组以及与其他调节因子靶基因位点结合的可能性。但是在构建海绵的过程中,只有一部分反义结合位点可以插入到载体中,目前尚未能够将所有内源性miRNA封闭[ 23]。
1. “封闭海绵”应用 miRNA海绵的直接应用是通过瞬时转染来获得所需的细胞类型。转染方法具有很大灵活性,转染时可选择不同启动子、载体、报告基因和miRNA靶基因结合位点来监测不同的细胞表型。最常用载体是质粒[ 13],也有人采用反转录病毒[ 14]、慢病毒[ 15, 16]进行转染。最常用的报告基因是绿色荧光蛋白报告基因[ 17, 18],还可以采用mcherry荧光蛋白基因或者萤光素酶报告基因。一般情况下,在转染海绵24~48 h后对细胞进行分析和靶基因验证。目前为止,独立的miRNA例如miR-155或大种子家族let-7都可用海绵成功封闭[ 10, 11]。
如果封闭成功,“封闭海绵”会导致miRNA水平特异性降低,在某些情况下Northern blot 也无法检测到miRNA[ 19]。这表明miRNA靶基因相互作用激发了miRNA降解。在果蝇和哺乳动物细胞内,当“封闭海绵”结合位点与miRNA的3′末端有充分结合时可加速miRNA核酸外切酶对miRNA的作用[ 20]。当构建的海绵有充足结合位点并含有可阻止“封闭海绵”本身降解的凸起时,上述情况更容易出现。另一个理想情况是在海绵中增加靶基因位点而不会导致miRNA表达水平上调。
早期细胞内瞬时转染海绵结果比较符合人们的预期效果,但由质粒介导的海绵瞬时转染只能满足对特定miRNA细胞水平监测,有时研究需要在体内长期稳定地封闭特定miRNA功能。 Gentner等[ 21]尝试用慢病毒载体把海绵序列稳定地过表达在细胞内并且成功地诱导出封闭特定miRNA功能的小鼠。Bhalala[ 25]通过封闭miR-21功能的小鼠研究得出miR-21在脊髓损伤中有着重要的调节功能。miR-574-5p 的封闭质粒帮助Li发现了其对TLR9的调控作用更深一步的揭露了miRNA与Toll样受体在肿瘤生物学之间的关联[ 26]。Wu等[ 27]构建了含有miR-20a海绵序列的质粒并用病毒载体将其表达在HEK-293T细胞中以此获得了稳定的细胞株系Jurkat-S。
2. “封闭海绵”应用中的缺陷 尽管miRNA海绵有很多优点,但其在不同环境中仍会表现出对miRNA程度不同的抑制。比如在miRNA浓度很高情况下,想要完全封闭其功能就需要很高的海绵剂量,而这个剂量可能难以达到,即该miRNA的封闭模型无法达到预期效果。换一个角度来讲,如果细胞中miRNA内源性靶基因很多,游离的miRNA就很少[ 22],这时则仅需要很少剂量海绵就能达到很好的封闭效果。
如果转入的海绵基因表达量很高(比如携带GFP报告基因)而又不确定其是否产生靶基因之外的效应时除了应在“封闭海绵”处理组与空载组间进行基因水平的对比,同时应与正常细胞组进行对比[ 10]。
确定转入的海绵是否能成功抑制目的miRNA比验证是否完整去除基因中成熟miRNA更具挑战性[ 7]。海绵效应可以通过验证已知靶基因的报告基因来检测,通常情况下采用融合在miRNA结合位点的荧光素酶报告基因或者一个已经确定的靶基因3′UTR来确定。在miRNA海绵抑制情况下荧光素酶报告基因表达水平明显降低[ 12]。但是由于报告基因受种子家族所有成员调控,所以这种情况下很难确定在种子家族不同成员中是否有相同抑制水平。
“封闭海绵”的发现为我们提供了研究miRNA功能的新方法,帮助我们在动植物及病毒中探索调节转录后水平新的未知角色[ 8]。随着越来越多非编码RNA的发现,它们的生物学功能有待验证,尤其是与miRNA存在的潜在关系。
综上所述,“封闭海绵”应用有着广阔前景,但由于还存在部分缺陷,所以对“封闭海绵”研究仍在不断继续和完善。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|


