对3种载脂蛋白E(
对518名健康调查者分别采用Multi-ARMS PCR、PCR-RFLP、PCR-SSCP等方法进行
在核酸序列分析结果一致性比较上,3种
PCR-RFLP在3种
To evaluate the 3 determination methods [multiplex amplification refractory mutation system polymerase chain reaction (Multi-ARMS PCR), polymerase chain reaction-restriction fragment length polymorphism (PCR-RFLP) and polymerase chain reaction-single strand conformation polymorphism (PCR-SSCP)] on the effect of apolipoprotein E (
The results of the 3 determination methods were not entirely consistent with the results of DNA sequencing (Multi-ARMS PCR
Among the 3 determination methods, PCR-RFLP is the most reliable one for
动脉粥样硬化(atherosclerosis,AS)是一种严重威胁人类健康的疾病,由动脉粥样硬化所致的冠心病(coronary artery heart disease, CHD)已成为西方国家以及一些东欧国家人口死亡的重要原因之一。我国动脉粥样硬化所致的死亡率已呈上升趋势。形成动脉粥样硬化的过程中,血脂代谢异常起着重要作用,载脂蛋白E (apolipoprotein E, apo E)是血浆脂蛋白的重要组成部分,在脂类的运输和代谢中起着十分重要的作用。 apo E的多态性取决于其第4外显子112位Cys/Arg (C112R)和158位Arg/Cys (R158C) 编码基因的多态性,3 种等位基因ε2、ε3 和ε4,可构成6 种不同的基因型:ε2/ 2、ε3/ 3、ε4/ 4、ε2/3、ε3/4 和ε2/4。不同等位基因与受体之间的亲和性有所不同,导致血脂代谢存在个体差异,例如ε4等位基因可以显著地升高健康人的总胆固醇浓度,使动脉粥样硬化发生危险性增加;相反,ε2等位基因可以降低胆固醇浓度,其降低效应是ε4升高胆固醇的2~3倍。因此 apo E基因型检测可以对此类疾病的一级预防提供有力的依据。 apo E基因型分析的方法很多,包括聚合酶链反应-限制性核酸内切酶片段长度多态性(polymerase chain reaction-restrition fragment length polymorphism, PCR-RFLP)、多重特异性扩增突变系统快速分型法(multiplex amplification refractory mutation system PCR, Multi-ARMS PCR)、聚合酶链反应-单链构象多态性(PCR-single strand conformation polymorphism, PCR-SSCPs)等。但各种方法结果准确度不一。我们以汉族健康人群作为研究对象,对3种分析 apo E基因型的方法的准确度进行探讨,试图为临床找出最准确有效的 apo E基因型检测的方法。
研究对象均为2011年6月至12月武汉大学中南医院的体检者,共518名,其中男305名,女性213名,年龄22~87岁,均为汉族。
采集研究对象空腹12 h以上的外周静脉血,乙二胺四乙酸(EDTA)抗凝。
TaqDNA聚合酶(美国Promega公司)、限制性核酸内切酶HhaⅠ(New Labs, England)Qiaquick PCR Purification Kit(美国Qiagen公司)、WV-BP330/G凝胶成像系统(江苏捷达公司)等。
1.Multi-ARMS PCR
采用改良NaI法提取外周血样本DNA作为模板。根据多重特异性扩增突变系统快速分型法(Multi-ARMS PCR)[ 1]进行引物设计:上游内引物P1(5'-ATGCCG ATGACCTGCAGAATT-3'),P2(5'- ATGCCGATG ACCTGCAGAATC-3');上游外引物P3(5'-CGC GGACATGGAGGACGTTT-3'),P4(5'-CGCGGACA TGGAGGACGTTC-3');下游引物P5(5'-GTT CAGTGATTGTCGCTGGGCA-3')。采用P1/ P2和P5扩增长度为451 bp的 apo E基因第4外显子的一段基因序列(覆盖编码第112、158位氨基酸的基因序列),采用P3/P4和P5扩增长度为588 bp的片段(同样覆盖编码第112、158位氨基酸的基因序列)。另外,为了保证扩增结果的可靠,在扩增体系中同时进行低密度脂蛋白受体外显子13上一段228 bp的片段作为内参,引物采用P6(5'-AACAACTGACCCCGCTGGCG-3'),P7(5'-ATGGCG-CTGAGGCCGCGCTC-3')。由上海生工合成。采用25 μL扩增体系A、B 2管,A管中包含200 ng模板DNA、20 pmol P5、15 pmol P1、10 pmol P3、dNTPs 200 μmol/L、MgCl2 1.5 mmol/L、DMSO 2.5 μL、TaqDNA聚合酶1 U;B管引物包含15 pmol P2、10 pmol P4、20 pmol P5,其余与A管一致。扩增条件为95 ℃预变性5 min;95 ℃变性45 s、62 ℃退火45 s、72 ℃延伸45 s,35个循环;最后72 ℃延伸7 min。PCR产物直接用含GoldviewTMDNA染料2%琼脂糖凝胶电泳鉴定。
2.PCR-RFLP
采用改良NaI法提取外周血样本DNA作为模板。扩增长度为292 bp的 apo E基因第4外显子的一段基因序列(覆盖编码第112、158位氨基酸的基因序列,设计引物如下:P8(5'-AACAACTGACCCCGGTGGCG-3');P9(5'-ATGGCGCTGAGGCCGCGCTC-3')[ 2]。由上海生工合成。采用25 μL扩增体系,包含200 ng模板DNA、20 pmol P5、30 pmol P8、30 pmol P9、dNTPs 200 μmol/L、MgCl2 1.5 mmol/L、DMSO 2.5 μL、TaqDNA聚合酶2 U。扩增条件为95 ℃预变性12 min;94 ℃变性1 min、65 ℃退火1 min、72 ℃延伸1.5 min 35个循环;最后72 ℃延伸15 min。PCR经含GoldviewTMDNA染料1.5%琼脂糖凝胶电泳鉴定。酶切PCR产物:20 μL酶切体系包含PCR产物8 μL,HhaⅠ内切酶2 U ;37 ℃水浴16 h。酶切产物经8%非变性聚丙烯酰胺凝胶电泳,银染显影鉴定。
3.PCR-SSCPs
采用改良NaI法提取外周血样本DNA作为模板。扩增长度为242 bp的 apo E基因第4外显子的一段基因序列(覆盖编码第112、158位氨基酸的基因序列)设计引物如下:P10(5'-ACAGAATTCGCCCCGGCCTGGTACAC-3');P11(5'-TAAGCTTGGCACGGCTGTCCAAGGA-3')[ 3]。由上海生工合成。扩增体积为25 μL,其中200 ng模板DNA、30 pmol P10、30 pmol P11、dNTPs 200 μmol/L、MgCl2 1.5 mmol/L、TaqDNA聚合酶2 U。扩增条件为95 ℃预变性5 min; 95 ℃变性1 min、60 ℃退火1 min、72 ℃延伸2 min,30个循环;最后72 ℃延伸10 min。PCR经含GoldviewTMDNA染料1.5%琼脂糖凝胶电泳鉴定。PCR产物经8%变性聚丙烯酰胺凝胶电泳,银染显影鉴定。
4.核酸序列分析
将所有样本采用PCR-RFLP方法中采用的扩增引物和条件进行扩增,纯化后由上海基康进行核酸序列分析。
采用SPSS 17.0 软件包分析。多行多列计数资料一致性分析采用 Kappa检验,并采用分型结果正确人数占受试总人数的百分率来计算各分型方法的准确度。
3种方法均检出 apo E 6种基因型(ε3/3、ε3/4、ε2/3、ε2/4、ε4/4、ε2/2),与核酸序列分析结果一致性比较显示,Multi-ARMS PCR Kappa值为0.964、PCR-RFLP为0.991、PCR-SSCPs为0.913( Kappa越接近1,提示一致性越好)。见表1、图1~3:
| 表1 3种方法检出 apo E各基因型频率分布[例(%)] |
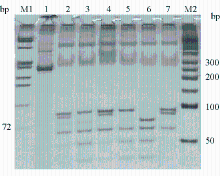
注:1为PCR产物(242 bp); 2为ε2/4(91+83+72+61+48+35 bp); 3为ε3/4(91+72+61+48+35 bp); 4为ε2/3(91+83+61+48+35 bp); 5为ε3/3(91+61+48+35 bp); 6为ε4/4(72+61+48+35 bp); 7为ε2/2(91+83+61 bp); M1为X174DNA/HaeⅢ markers; M2为100 bp ladder
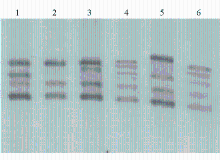
注:1为ε3/3; 2为ε2/2; 3为ε2/3; 4为ε3/4; 5为ε4/4; 6为ε2/4;电泳图上未出现未被变性的双链及变性后又复性的双链条带
注:M为100 bp ladder; 1、2, 3、4, 5、6, 7、8, 9、10, 11、12泳道为6种基因型A、B管电泳图谱(从左到右分别为ε3/3、ε4/4、ε3/4、ε2/2、ε2/3、ε2/4); 228 bp处条带为LDLR内参PCR产物
以核酸序列分析结果为金标准,与测序结果一致的认定为分型结果正确,与测序结果不一致的认定为分型错误,用分型结果正确人数占受试总人数的百分率表示准确度。结果显示PCR- RFLP准确度最高(99.8 %)、Multi-ARMS PCR准确度为99.2%%、PCR-SSCPs为98.5%。见表2:
| 表2 3种 apo E基因分型法准确度评价表 |
Multi-ARMS、PCR-RFLP、PCR-SSCP 3种方法与核酸序列分析结果一致性分析(以 Kappa值表示)来看,PCR-RFLP结果的一致性最好( Kappa=0.991, P<0.01),Multi-ARMS次之( Kappa=0.964, P<0.01),PCR-SSCP较差( Kappa=0.913, P<0.01),提示PCR-RFLP与核酸序列分析结果一致性最高,准确度达99.8%,因而作为一种 apo E基因分型的常用方法[ 2, 4, 5, 6, 7, 8, 9, 10],优点包括PCR条件成熟、电泳结果染色后条带位置、数目稳定,结果观察清楚;但此法步骤较繁琐,用时较长,不适用于临床常规检查,可作为实验室研究方法及临床确证方法。
Multi-ARMS法的结果准确度较PCR-RFLP略差(99.2%),但步骤相对较为简单,直接提取模板DNA、经基因扩增后可直接进行琼脂糖凝胶电泳得出结果,可作为一种快速、简便的方法推荐到临床作为常规 apo E基因分型的方法,但还是有一定的判读错误率,主要是在PCR后模板的扩增量不足至琼脂糖凝胶电泳时DNA条带的荧光强度不够、条带较模糊的情况下,容易出现结果判断不准确,这时采用准确性更高的方法(如PCR-RFLP法、核酸序列分析)进行验证。
PCR-SSCPs在3种方法中准确度相对较差(98.5%),方法也较繁琐,PCR产物片段大小、模板DNA的变性程度、凝胶浓度、染色效果等因素会直接影响条带的数目、位置和变性聚丙烯酰胺凝胶电泳的分辨力,在 apo E基因分型上没有明显优势,但可以作为 apo E未知突变位点的筛查方法。
本研究结果表明,在 apo E基因分型的3种方法中,准确度最高的是PCR-RFLP法,可作为临床 apo E基因分型的确证首选方法; 而Multi-ARMS由于其检测时间短、操作简单等优点可作为的一种快速的筛查方法推荐给临床应用。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|