通讯作者:蒋燕群,联系电话:021-64369181-8848。
作者简介:王欣慧,女,1984年生,硕士,主要从事细菌耐药性研究。
检测产AmpC β-内酰胺酶(简称AmpC酶)的大肠埃希菌中编码非必需青霉素结合蛋白4 (PBP4)的基因 dacB mRNA表达水平,探讨PBP4在产AmpC酶的革兰阴性菌耐药机制中的作用。
方法收集上海市第六人民医院2003至2010年间部分产AmpC酶的大肠埃希菌34株,聚合酶链反应(PCR)扩增 dacB基因和 ampC基因,实时定量PCR检测 dacB mRNA表达水平,并分为 dacB mRNA表达上调组与 dacB mRNA表达下调组,实时定量PCR方法检测 ampC mRNA表达水平。微量肉汤稀释法药敏试验检测抗菌药物的敏感性。
结果34株大肠埃希菌中,26株(76.47%) dacB mRNA表达水平上调, dacB mRNA表达水平上调组 ampC mRNA表达水平高于下调组, P<0.05。
结论推测大肠埃希菌临床菌株中PBP4高表达有助于产生AmpC酶的高表达,从而引起细菌耐药性的增加。
To detect the mRNA expression level of the nonessential penicillin-binding protein 4 (PBP4) dacB gene in AmpC beta-lactamase producing Escherichia coli isolates,and investigate the effect of PBP4 in the resistant mechanism of AmpC beta-lactamase producing gram-negative isolates.
MethodsA total of 34 AmpC beta-lactamase producing Escherichia coli isolates were collected in Shanghai Sixth People's Hospital from 2003 to 2010.The polymerase chain reaction(PCR) was used for the amplification of dacB gene and ampC gene, and the real-time quantitation PCR was used for the detection of the mRNA expression level of dacB. The isolates were classified into 2 groups: the high expression level group and the low expression level group. Real-time quantitation PCR was used for the detection of the mRNA expression level of ampC. The broth microdilution method was used for the detection of sensitivity.
ResultsAmong the 34 Escherichia coli,26 (76.47%) of 34 isolates had mRNA overexpression level of dacB,and the mRNA of ampC in the high expression level group was higher than that in the low expression level group( P<0.05).
ConclusionsThe overexpression of PBP4 may be helpful to introduce high-level resistance by triggering the overexpression of AmpC beta-lactamase among clinical isolates of Escherichia coli.
临床上引起各种感染的肠杆菌科细菌中, 大肠埃希菌是最常见的细菌之一。通过各种耐药机制而引起其耐药菌株的流行, 降低了治疗感染性疾病的疗效, 这将会严重威胁到医疗安全[1]。近年来从上海市第六人民医院耐药监测结果来看, 大肠埃希菌一直保持着最高的分离率, 且耐药率也逐年增加。在细菌耐药机制研究中, 发现酶的水解作用是耐药增加的一个重要原因。β -内酰胺酶一直是关注的焦点, 其中AmpC酶对大量抗菌药物产生耐药, 包括青霉素和除头孢匹罗和头孢吡肟以外几乎全部的广谱头孢菌素[2]。产ESBLs和AmpC酶的大肠埃希菌不但会导致治疗的失败, 而且通常还会携带其他的耐药基因, 这也限定了对其治疗的选择[3]。AmpC酶的产生是一个由多个部分参与的复杂的过程, 研究显示, 许多参与诱导的基因也参与了菌体细胞壁的循环过程。本研究以产AmpC酶的大肠埃希菌为研究目标, 探讨这些细菌中青霉素结合蛋白4(PBP4)表达情况及其在耐药中的作用。
1. 菌株 收集上海市第六人民医院2003年1月至2010年6月痰、尿等各种临床标本中分离的大肠埃希菌, 并经头孢西丁初筛试验、三维试验和多重聚合酶链反应(PCR)试验筛选出34株产AmpC酶的菌株。
2. 试剂 药敏试验板购自上海复星佰珞生物技术有限公司; PCR扩增试剂盒、逆转录PCR(RT-PCR)反应试剂盒、实时定量PCR试剂盒均购自大连宝生物工程有限公司; 细菌总RNA提取试剂盒购自天根生物有限公司; 引物由上海生工和大连宝生物工程有限公司合成。
3. 仪器 WALKAWAY-40微生物鉴定药敏系统为美国DADE-BERING公司, 基因扩增仪、实时荧光定量PCR仪为罗氏公司。
1. 三维试验 参照文献[4], 通过三维试验的方法检测AmpC酶。操作步骤主要包括:(1)反复冻融法制备酶粗提取物; (2)头孢西丁纸片贴于涂布有大肠埃希菌(ATCC 25922)的水解酪蛋白(MH)琼脂平板中央, 并用无菌刀片沿纸片边缘5 mm处放射状切一裂缝; (3)酶粗提物加入切裂缝内; (4)35 ℃孵育16~18 h。
2. 多重PCR试验 热裂解法提取基因组DNA, 配制反应体系50 μ L, PCR反应程序为94 ℃ 5 min, 94 ℃ 30 s, 64 ℃ 30 s, 72 ℃ 1 min, 30个循环, 72 ℃ 7 min[5]。
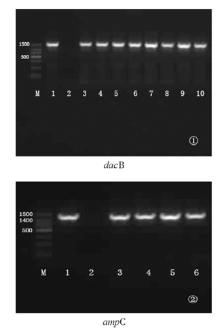
3. PCR检测dacB和ampC基因 热裂解法提取细菌DNA模板, 配制反应体系50 μ L, 引物见表1。 dacB和ampC PCR反应条件均为94 ℃ 5 min, 94 ℃ 30 s、60 ℃ 30 s、72 ℃ 60 s 30个循环, 72 ℃延伸10 min; 产物长度分别为1 216bp和1 061bp。
| 表1 dacB PCR和RT-PCR引物 |
4. 实时定量PCR试验 实时定量 PCR检测dacB和ampC mRNA表达水平, 引物见表1。(1)菌株总RNA的提取:按照天根生物有限公司细菌总RNA提取试剂盒的说明书进行操作, 主要步骤包括, 收集菌体, 裂解细菌, 去除蛋白质和DNA, 检测收集的总RNA纯度及浓度; (2)逆转录:将RNA逆转录为cDNA, 按照试剂盒说明书配制10 μ L反应体系, 反应条件为37 ℃ 15 min, 85 ℃ 5 s, 反应产物作为实时定量PCR的模板; (3)实时定量PCR:参照说明书配制20 μ L反应溶液, 反应程序均为95 ℃ 30 s 1个循环; 95 ℃ 5 s、60 ℃ 20 s、72 ℃ 15 s 40个循环。实验重复3次取均值, 以16s rRNA为内参; (4)以大肠埃希菌ATCC 25922为标准对照:参照Kenneth等[6]推荐的方法进行分析。以标准菌株的dacB和ampC mRNA表达水平为基准, 临床菌株与其比较。其中, 域值循环差(Δ CT)=目的基因CT值-内参CT值, 标本域值循环差(Δ Δ CT)=临床菌株目的基因CT值-标准菌株目的基因CT值; 2-Δ Δ CT即为与标准菌株相比cDNA的含量。
5. 药敏试验 采用微量肉汤稀释试验测定药物的最低抑菌浓度(MIC)值, 并严格按照美国临床实验室标准化协会(CLSI)2011年制定的规则及标准判定结果。
一、三维试验阳性的111株菌经多重PCR反应、琼脂糖凝胶电泳出现阳性条带的共34株。
二、PCR扩增dacB和ampC基因结果显示, 实验菌株均为阳性, 见图2。从dacB基因测序结果来看, 并未见各种原因而致基因缺失。
三、实时定量PCR检测所有大肠埃希菌dacB mRNA表达水平大肠埃希菌26株PBP4表达水平上调, 占76.47%, 同时检测ampC基因的表达水平, 结果见表2。对ampC 2-Δ Δ CT结果进行t检验, 上调组ampC2-Δ Δ CT均值为3.86, 下调组ampC 2-Δ Δ CT均值为0.69, 检测结果差异有统计学意义(P< 0.05), 即在dacB mRNA表达水平上调组中的ampC的表达水平高于下调组dacB mRNA表达水平。
| 表2 dacB、ampC基因实时定量PCR检测结果 |
四、药敏试验结果
微量肉汤稀释试验检测所有菌株对8种抗菌药物敏感性, 敏感率见表3。结果显示, 本实验菌株中dacB mRNA表达上调组与dacB mRNA表达下调组对一代、二代和三代头孢菌素均不敏感, 亚胺培南、氨基糖苷类药物阿米卡星和氟喹诺酮类药物环丙沙星敏感率相对降低。
| 表3 8种抗菌药物的敏感率(%) |
在我院临床分离到的各种致病菌中, 大肠埃希菌分离率最高, 同时在临床上, 其也是引起各种感染中最常见的革兰阴性杆菌。抗菌药物的高选择性使细菌产生了各种各样的变异, 耐药性不断增加, 细菌产酶引起的耐药被认为是主要耐药机制。以往, 一直是在革兰阳性细菌的耐药机制中研究PBP4, 却忽视了临床中出现的革兰阴性杆菌耐药机制中PBP4的重要作用。近年来学者们认识到在革兰阴性杆菌耐药机制中, PBP4也起到了重要的作用, 其中非必需PBP4的作用正越来越受到关注。本研究旨在探讨产AmpC酶的大肠埃希菌临床菌株中PBP4所起作用。
本研究所选菌株均产AmpC酶, 在产AmpC酶的相同背景下区分出PBP4上调组与PBP4下调组, 目的在于同为产AmpC酶的情况下比较PBP4在耐药中所起的作用。E.coliK-12编码了13种[7]功能性的PBP4, 根据与细菌生理功能的关系大致可分为细菌生长必需PBP4和非必需PBP4。尽管非必需PBP4不是细菌生存所必需, 但其在菌体内又保持着高的拷贝量, 这说明在菌体生存中PBP4除了维持细胞形态外可能还具有其他的生理功能, 所以PBP4作为本研究的关注对象。在筛选出产AmpC酶的大肠埃希菌中dacB mRNA表达上调菌高达76.47%, 且通过比较分析编码PBP4基因dacB mRNA表达上调组与下调组对抗菌药物的敏感率, 结果显示对于一代、二代和三代头孢菌素均不敏感, 所以PBP4作用无法通过这3种抗菌药物的敏感率体现出来。而在头孢吡肟、亚胺培南、阿米卡星、环丙沙星中dacB mRNA表达上调组敏感率均相对低于下调组, 提示PBP4可能耐药中起到一定的作用。
本研究在国内首次对临床菌株进行了dacB基因的检测, 分析其与耐药的相关性。Moya等[8]提出铜绿假单胞菌中, dacB失活使AmpC酶高表达从而增加了其对β -内酰胺类抗菌药物的耐药性。通过本研究对ampC检测和药敏分析结果显示, dacB mRNA表达水平上调组的ampC表达水平普遍高于下调组, 经t检验证实差异具有统计学意义。推测大肠埃希菌中dacB mRNA高表达可能通过促进ampC的表达而提高AmpC酶的产生, 从而降低对β -内酰胺类抗菌药物敏感性, 但是PBP4所起的作用还需要进一步实验证明。目前对PBP4在革兰阴性杆菌中作用的研究, 使我们进一步理解了β -内酰胺类抗菌药物产生耐药的机制, 同时希望在感染性疾病的治疗中, 能从新的角度寻找抗菌药物的作用目标。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|