作者简介:王志林,男,1972年生,副主任技师,主要从事白血病遗传学研究。
慢性粒细胞白血病(CML)是一种起源于多能造血干细胞的克隆性恶性血液病。其细胞遗传学特征是9号和22号染色体的相互易位,即Ph染色体。该易位导致BCR/ABL融合基因,后者在CML的发病中起着极其重要的作用。最近,荧光原位杂交(FISH)技术证实约10%~15%的CML患者在衍生9号染色体[der(9)]易位断裂点附近有较大的序列缺失。研究表明[ 1],伴有[der(9)]部分序列缺失的患者有较短的慢性期,较快的疾病进展速度及较短的生存期,因而认为[der(9)]部分序列缺失是一项强有力的预后指标。我们发现1例CML加速期患者,染色体核型正常,FISH发现患者存在[der(9)]而衍生22号染色体[der(22)]缺失。
1. 研究对象 患者男,59岁,2005年4月在常州二院诊断为CML,核型为46,XY,t(9;22)(q34;q11),患者一直用羟基脲和干扰素治疗;2009年6月在常州二院复查,CML慢性期,核型为46,XY[19]/46,XY,t(9;22)(q34;q11)[1],2个月前双足溃疡,2009年月10月13日入常州市第一人民医院治疗,外周血:白细胞(WBC):12.1×109/L,血红蛋白(Hb):117 g/L,血小板(PLT):912×109/L。
骨髓涂片显示:骨髓增生明显活跃,其中粒系占87.0%,红系占11.0%,粒红比例为7.91∶1,粒系异常增生,以中幼粒以上阶段为主,各期细胞胞体较大,原始细胞+早幼粒细胞为25.5%,呈圆形或类圆形,核部分不规则,可见巨杆状粒细胞,易见嗜酸、嗜碱粒细胞,嗜酸细胞占5.0%,嗜碱细胞占2.0%。红系增生以中晚红为主,淋巴细胞占2.0%。全片巨核细胞大于1 000个,巨核细胞胞体小,易见小巨核细胞,可见小巨核细胞增生灶,PLT明显增多,成簇成堆多见。诊断:CML进展期。
2. 染色体核型分析 采用骨髓细胞短期培养法,按常规制备染色体并进行R显带处理。核型按《人类细胞遗传学国际命名体制(ISCN,1995)》加以描述。
3. 双色荧光原位杂交(D-FISH)检测 试剂为北京金菩嘉医疗科技有限公司,采用SpectrumOrange标记的序列特异性DNA探针ABL(GLP ABL),以及SpectrumGreen标记的序列特异性DNA探针BCR(GLP BCR)。
取出于-20 ℃储存患者染色体标本,换用新鲜的甲醇/冰醋酸(3∶1)固定液,气干法滴片,隔夜老化,置于常温2×SSC中脱水5 min,在70%、85%、100%室温乙醇中梯度脱水各2 min,晾干。探针处理:每份标本探针1 μL,加杂交稀释液4 μL混匀,将探针混合液5 μL加于玻片的待杂交区,盖上盖玻片。用橡胶胶水(Rubber Cement)封片后,置于自动原位杂交仪(ThermoBrite)中,程序设定78 ℃变性5 min,42 ℃杂交20 h。杂交结束后,洗片,去除盖玻片,将玻片置于46 ℃的0.3%NP-40/0.4×SSC中洗涤2 min,再置于室温2×SSC /0.1NP-40中洗涤45s(NP-壬基酚聚氧乙烯醚),置于2×SSC中脱水4 min,再置于70%的酒精中脱水4 min,取出玻片,晾干,复染,吸取 10 μL的4,6-二脒基-2-苯基吲哚(DAPI)于杂交区,置于4 ℃冰箱,待30 min后观察结果。
检测:用OLYMPUS BX51正置荧光显微镜在DAPI/FITC/RHOD滤光片的激发下,观察细胞中桔红色和绿色荧光杂交信号,每例至少分析200个细胞,计数边界清楚,无重叠,结构完整的细胞核,用德国耶拿CCD摄像头进行图像采集,使用IMSTAR软件进行图像分析。
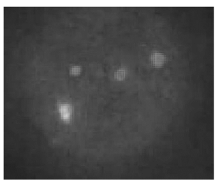
1. 染色体核型分析 常规染色体R带核型分析显示患者的骨髓细胞核型为46,XY[20],没有发现有Ph染色体,见 图1。
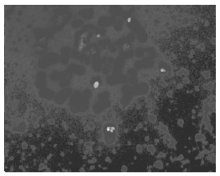
2. D-FISH检测 每一个细胞都有一个BCR和ABL融合信号,见 图2,以为是der(9)缺失,但该融合信号不在22号染色体上,而在9号染色体上,患者是der(22)缺失,见 图3。
CML患者接受羟基脲和干扰素的联合化疗,或格列卫治疗,与此同时,患者在获得遗传学缓解期间出现的Ph阴性异常克隆演变(Ph-CE)也逐渐被关注[ 2]。国内有学者[ 3]观察的伊马替尼治疗有效的100例CML各期患者中有11例(11%)被检出Ph-CE,发生率与文献报道相近,异常核型亦以+8,+Y为常见,Ph-CE可见于Ph+克隆开始减少或完全消失后。
在我们的研究中,患者存在[der(9)]而[der(22)]缺失。患者出现[der(22)]缺失是不是Ph阴性异常克隆演变,9号染色体上的ABL/BCR融合基因基因究竟起什么作用,有待进一步研究。为什么Ph染色体会消失,而患者处于疾病加速期。总之,肿瘤发病机制还有待进一步探讨。
| [1] |
|
| [2] |
|
| [3] |
|