作者简介:项明洁,女,1967年生,硕士,主任技师,主要从事临床微生物学与免疫学检验、教学和科研工作。
运用Agilent生物分析仪对临床白假丝酵母菌进行重复序列(REP)-聚合酶链反应(PCR)分型,并分析其流行趋势。
方法设计REP引物,进行PCR扩增,用DL7500 Labchip芯片在Agilent 2100 生物分析仪上对产物做微流电泳,获得虚拟凝胶视图,再运用GelCompareⅡ 软件对其DNA指纹图谱做聚类分析。
结果白假丝酵母菌菌株指纹图谱相似度较高,>80%以上。被分析的100株临床株可分为9个基因型。
结论REP-PCR的Agilent 2100同源性分析具有快速、易于操作、高重复性和高分辨率等优点,可广泛用于流行病学调查与研究。
To apply Agilent bioanalyzer in genotyping of clinical Candida albicans by repetitive element (REP)-polymerase chain reaction(PCR)and analyze the epidemic trends.
MethodsPCR amplification was performed by REP as primers, and the micro-flow electrophoresis of its amplicons was conducted on DL7500 Labchip by Agilent 2100 bioanalyzer. The virtual gel view was gotten. The cluster analysis of virtual gel view of DNA fingerprinting was used by GelComparⅡ software.
ResultsDNA fingerprinting similarity of Candida albicans strains was high and more than 80%. 9 genotypes were found from 100 clinical analyzed strains.
ConclusionsREP-PCR of Agilent 2100 bioanalyzer can be used for epidemiological investigation and research with the advantages of rapid, feasible operating, high reproducibility and high resolving power.
白假丝酵母菌感染已成为获得性免疫缺陷综合征、肿瘤、器官移植、自身免疫病等免疫缺陷患者的死亡原因之一。该菌也是医院感染的常见病原菌, 可引起爆发流行。为预防和控制医院感染的流行传播, 其基因型别分析及流行病调查研究尤为重要。脉冲场凝胶电泳(PFGE), 其操作流程和规则均有参照, 较其他一些非标准化检测方法可比性强、重复性佳。本研究运用重复序列(REP)-聚合酶链反应(PCR)分型技术对白假丝酵母菌进行基因型别扩增, 并用Agilent 2100生物分析仪对扩增产物进行微流电泳, 比对其DNA指纹图, 以了解该菌在院内的流行情况。
1. 实验菌株 本研究共纳入白假丝酵母菌100株, 其中来自上海市瑞金医院微生物科2009年1月至7月临床分离并保存的菌株92株, 来自上海市第一妇婴保健院菌株8株。所获临床分离株分别为痰标本64株, 阴道分泌物9株, 粪便7株, 鼻拭子6株, 尿液5株, 深静脉导管3株, 咽喉拭4株, 胆汁2株。平行选择白假丝酵母菌质控敏感株ATCC 90028 1株和临床分离对照菌光滑假丝酵母菌1株、热带假丝酵母菌1株、近平滑假丝酵母菌2株、克柔假丝酵母菌2株。
2. 仪器与试剂 BIOER TC-96-T/HA PCR扩增仪; Agilent 2100 生物分析仪; DL7500 Labchip芯片购自法国生物梅里埃公司; 随机引物及Taq多聚酶购自上海生工生物工程有限公司; 破壁采用的裂解剂Lyticase为Sigma公司产品。
1. 菌种鉴定 所有菌株均经酵母菌显色培养基、芽管试验、厚壁孢子试验和法国生物梅里埃的API试条鉴定确认。
2. REP-PCR分型 (1)DNA提取:挑取单个菌落, 溶解于100 μ L裂解液中, 置沸水浴3040 min, 12 000 r/min(离心半径为5 cm)离心5 min, 收集上清液; (2)PCR扩增:应用REP-PCR引物进行扩增, 反应体系25 μ L[10× 缓冲液 2.5 μ L; Mg2+ (3 mmol/L)3 μ L, 2.5 μ mol/L dNTP 0.2 μ L, 引物(10 μ mol/L)0.1 μ L, 模板4 μ L, Taq DNA聚合酶1 μ L]。引物序列以Care-2 重复元件设计[1], Ca-21 为5'-CATCTGTGGTGGAAAGTT-AAC-3', Ca-22为5'-ATAATGCTCAAAGGTGGT-AAG-3'。循环条件为:94 ℃ 7 min预变性, 94 ℃变性1 min, 52 ℃退火1 min, 65 ℃延伸8 min, 65 ℃终止16 min, 重复35个循环; (3)Agilent 2100 生物分析仪分析:具体操作参见“ Agilent DNA 7500 and DNA 12000 kit Guide” , 其检测原理为Ca-21和Ca-22引物与基因组DNA在相应部位结合, 从而产生各种片段大小不一的扩增产物, 扩增的片段在DNA芯片装置下由微流体分开, 荧光吸收检测后分析仪收集并分析数据, 同时产生相应的峰值图, 不同峰值代表不同大小DNA片段, 并形成虚拟凝胶视图的DNA指纹图谱。
应用GelComparⅡ 软件(Applied Maths)对虚拟凝胶视图的DNA指纹图谱做聚类分析。Dice相关系数分析指纹图谱相似度, UPGMA得到单匹配系数矩阵图, 并生成表示菌株之间相似性的树状图。
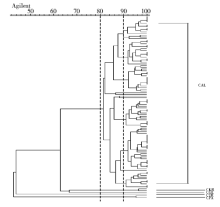
本研究以Ca-21、Ca-22 1对引物所扩增的产物, 通过Agilent 2100微流电泳产生稳定、清晰的指纹图谱。假丝酵母菌属PCR扩增产物大小多为601 800 bp, 条带数为7条以上, 白假丝酵母菌条带较多, 约15条, 与非白假丝酵母菌DNA扩增带型有明显区别, 见图1、2。聚类分型结果显示, 100株临床分离的白假丝酵母菌, 相似度较高, > 80%以上, 在树状图相似系数(SAB)=90%相似度水平上, DNA带型可被分为9型: 26株为A型, 16株为B型, 16株为C型, 15株为D型, 10株为E型, 14株为F型, G、H、I型各为1株。见图1、3、4。
根据所测得分离株的不同基因型(AI), 结合分离菌株的标本来源, 将其具体分布情况列表显示, 见表1。虽未能发现有特殊型集中性分布现象, 但对上海市第一妇婴保健院的8株均为同型应值得关注。
| 表1 白假丝酵母菌瑞金医院各病区及上海市第一妇婴保健院菌株型别分布情况 |
近年来, 随着肿瘤、器官移植、糖尿病等患者不断增多, 相关侵入性诊断治疗手段的广泛实施以及各类免疫抑制剂和广谱抗菌药物的大量使用, 使得临床上条件致病真菌感染明显上升。有资料显示, 深部真菌感染病例中70%80%是由白假丝酵母菌感染引起的, 并伴随着一定数量的耐药株出现。为防止其在院内大量免疫力低下患者中的传染流行, 有必要对该菌基因型别及流行趋势进行调查分析, 便于掌握院内流行的可能性趋势, 力求做好有效的预防和治疗。
REP-PCR指纹分析技术是由Versalovic等[2]90年代初首次运用于病原微生物的研究, 是一种基于病原体基因组内存在短的重复序列而建立起来的新型的微生物分型技术。由于其具有高分辨力、简单易行和良好的可再现性等特点, 已广泛地应用于多种生物医学研究领域[3, 4]。REP-PCR分型技术所用的Agilent 2100生物分析仪是一种标准化的自动化系统, 分析时只需1 μ L的微量DNA模板, 12个标本40 min出结果, 具有很高的分辨率, 重复性好, 操作简单[57]。分析数据以电子文档的形式存盘, 对不同批次和不同实验室标本具有同样的可比性; 同时其结果既有虚拟凝胶电泳图输出形式, 也可以荧光强度直方图形式输出, 对条带荧光强度进行量化。
本研究运用Care-2重复序列设计引物, 对所采集到的临床白假丝酵母菌做相关基因扩增, 并通过Agilent 2100生物分析仪和GelComparⅡ 软件进行型别分析, 以期获得目前有关临床分离白假丝酵母菌流行情况的资料, 并评价该REP-PCR对白假丝酵母菌基因分型的可行性。
虚拟凝胶电泳图及荧光强度直方图显示白假丝酵母菌条带较多, 明显区别于非白假丝酵母菌。聚类分型结果显示, 假丝酵母菌属种间差异较大, 白假丝酵母菌与非白假丝酵母菌相似度< 70%, 提示该分型技术的种间鉴别能力佳。Ca-21、Ca-22引物对100株临床分离的白假丝酵母菌种内分型, 种内相似度较高, > 80%以上, 分为9型, 其中主要为6型, 3株各成一型, 为少见株, 说明该分型技术具有较强的分辨率和分型力。基因型别结果显示, 采自上海市第一妇婴保健院8株菌为同一型别— — B型, 可能有院内感染流行的发生, 但样本量较小, 需要密切关注。而上海市瑞金医院则并未发现型别分布有明显倾向性, 其基因型别呈现多样性, 说明白假丝酵母菌未在该院形成院内流行。进一步对院内不同病区的分离株型别进行分析, 发现呼吸科八病区感染菌株主要集中在A型和D型, 肾脏病区和九舍病区主要分别为F型、C型, 呈相对集中趋势, 而其他科室型别分布较为分散。提示院内的各病区间尚未形成流行株, 但对个别特殊病区应当预防内部流行的发生。
追踪感染源、调查传染途径是分型技术的意义所在, 本研究收集的100株临床白假丝酵母菌株主要来自于上海市瑞金医院, 存在地域和数量的局限性, 需要后续加大样本量, 扩大调查范围(包括不同医院、病区间护理工、公用仪器等), 使得REP-PCR分型技术能更为客观地反映白假丝酵母菌院内流行、病区流行情况, 在明确感染源及传播途径上发挥更大作用。同时比对不同耐药谱菌株的REP-PCR指纹图谱, 是否能够有助于了解耐药株的特定指纹图谱的存在尚待研究。
总之, REP-PCR分型技术联合Agilent 2100生物分析仪具有快速、易于操作、高重复性和高分辨率, 更为关键的是可对不同批次和不同实验室结果具有一致的可比性等优点, 可作为分型鉴定的有效工具, 尤其是在缺乏专业仪器设备的实验室(如PFGE技术和DNA测序)以及短期内疾病爆发流行大样本检测时, 可作为首选技术。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|