作者简介:汤瑾,女,1975年生,硕士,主管技师,主要从事细菌耐药监测与研究。
通讯作者:蒋燕群,联系电话:021-64369181-58735。
1998年Fire等[1]首次对秀丽线虫(Caenor-habditis elegans)注射双链RNA(dsRNA)时发现, dsRNA能够引起与该段RNA同源的mRNA产生特异性降解, 从而高效地特异性阻断相应基因的表达, 他们把这种发生在转录后的基因沉默(post-transcriptional gene silencing, PTGS)现象命名为RNA干扰(RNA interference)。这一现象广泛存在于自然界各级生物体中, 是生物体清除病毒等外源性核酸或抑制突变及异常内源性基因表达的一种自我保护机制[2], 他的出现为基因功能研究提供了一种新的具有高效性和高度特异性的功能基因组研究策略。目前RNA干扰技术主要用于对病毒感染(肝炎病毒和人类免疫缺陷病毒)和肿瘤等疾病的研究, 抑制病毒基因的表达、复制以及肿瘤生长。2006年, 日本学者首次用双链小RNA(SiRNA)诱导了耐甲氧西林金黄色葡萄球菌(MRSA)凝固酶基因的沉默, 其mRNA转录水平下降, 凝固酶合成受阻, 动物模型显示小鼠的感染好转[3], 这是RNA干扰技术首次运用在细菌基因的研究。我们运用RNA干扰技术探讨blaKPC表达与碳青霉烯类耐药的关系。
1.研究对象 产肺炎克雷伯菌碳青霉烯酶-2(KPC-2)的肺炎克雷伯菌(A1500)分离自上海瑞金医院微生物科, 产KPC-2的肺炎克雷伯菌(K1)分离自浙江大学附属第二人民医院检验科。质控菌株为大肠埃希菌(ATCC 25922), 购自上海市临床检验中心。
2.SiRNA的设计与合成 以blaKPC-2 (基因号EU594573)为目标基因, 表达量相对稳定的肺炎克雷伯菌16S rRNA基因(基因号FM207545)为管家基因, 肺炎克雷伯菌中不携带的凝固酶基因(基因号EU490370)为非特异性对照基因, SiRNA由上海英骏(invitrogen)生物有限公司设计与合成。 (1)针对目标基因blaKPC-2 (基因号EU594573) 的347~367、558~578、584~604碱基位设计3对21nt的SiRNA; (2)针对管家基因16S rRNA(基因号FM207545)的100~120、481~501碱基位设计2对21nt的SiRNA; (3)针对非特异性对照基因金黄色葡萄球菌凝固酶基因(基因号EU490370)的76~96碱基位设计1对21nt的SiRNA。见表1。
| 表1 SiRNA序列 |
3. 药敏纸条 亚胺培南、厄他培南E-test试验条购自广州乐通泰公司。
4. 电转杯 购自上海Bio-Rad公司。
5. 逆转录聚合酶链反应(RT-PCR)引物 由上海生工生物技术公司合成。见表2。
| 表2 RT-PCR引物序列 |
1.细菌培养 肺炎克雷伯菌(A1500)、K1接种在LB培养基上, 35 ℃培养16~18 h。
2.稀释 SiRNA加RNAase-free水稀释成20 μ mol/L备用, -20 ℃低温保存。
3.电转移 经过反复试验优化试验条件后, 104 cfu/mL的菌液400 μ L, 加SiRNA至最终浓度2 μ mol/L, 以电压1 200 V、脉冲时间2 ms共进行4次电转移。电转移后菌液移入LB肉汤, 35 ℃培养16~18 h。35 ℃孵育约18 h。
4.抽提mRNA(Tiangen总RNA抽提试剂盒) 抽提肺炎克雷伯菌(A1500)、K1的mRNA。
5.干扰效果检测 RT-PCR。(1)逆转录(20 μ L体系):dNTP(10 mmol/L)1 μ L, 随机引物(20 mmol/L)1 μ L, 模板(mRNA)500 ng, 焦碳酸二乙酯(DEPC)水至10 μ L; 65 ℃ 5 min, 离心数秒。5× primescript缓冲液 4 μ L, RNA inhibitor(40 U/μ L) 0.5 μ L, primescript RTase 0.5 μ L, DEPC水 5 μ L, 30 ℃ 10 min, 42 ℃ 20 min, 95 ℃ 5 min; (2)PCR扩增(50 μ L体系):10× 缓冲液5 μ L, dNTP(10 mmol/L)2 μ L, prime-F 0.5 μ L, prime-R 0.5 μ L, TaKaRa EX TaqHS(5 μ L/μ L) 0.5 μ L, 反转录液 4 μ L, DEPC水 37.5 μ L, 模板 (cDNA) 1 μ L; 95 ℃ 5 min后, 94 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 1 min 30个循环, 72 ℃ 10 min; (3)电泳:1.2%的琼脂糖凝胶电泳, 100 V, 120 mA, 30 min。
6.SiRNA干扰前后的肺炎克雷伯菌(A1500)、K1的体外药敏检测 将电转移前后的A1500、K1菌液35 ℃孵育至0.5麦氏后, 均匀涂布于水解酪蛋白胨(MH)琼脂平皿, 分别贴亚胺培南和厄他培南E-test纸条, 以大肠埃希菌(ATCC 25922)为质控菌株。
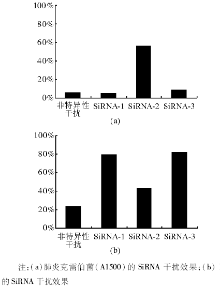
通过干扰管家基因16S rRNA进行电转移试验条件优化后, 再对目标基因blaKPC-2进行电转移, 根据干扰前后RT-PCR显示的目标条带深浅度判断干扰效果。继续做RT-实时PCR, 对干扰效率进行定量检测。结果显示肺炎克雷伯菌(A1500)的blaKPC-2干扰前后的效果不明显, K1的干扰效果要高于肺炎克雷伯菌(A1500)。K1中, SiRNA-1和SiRNA-3的干扰效果要高于SiRNA-2。见表3、图1。
| 表3 实时PCR计算SiRNA的干扰效率 |
SiRNA干扰虽然在RNA水平上抑制了blaKPC-2的表达, 但干扰前后菌株对亚胺培南和厄他培南的最低抑菌浓度(MIC)均为32 μ g/mL。
RNA干扰作为一种简单、有效的代替基因敲除的分子生物学手段, 加快了功能基因组学领域的研究步伐, 广泛应用于病毒性肝炎和肿瘤免疫的研究中[4~8]。RNA干扰用于细菌的基因功能研究是从2006年日本学者运用SiRNA抑制MRSA凝固酶的基因开始[3], SiRNA干扰了凝固酶基因mRNA转录水平, 凝固酶合成受阻, 动物模型显示小鼠感染好转[3, 4]。我们通过SiRNA对肺炎克雷伯菌的blaKPC-2进行干扰, 也证实了RNA干扰技术可以用于细菌基因功能的研究。
针对目的基因的不同序列合成的几组SiRNA的干扰效果具有随机性, 其干扰效果不能预测, 本研究针对blaKPC-2基因设计了3组SiRNA, 其干扰效果差异明显; 即使同一SiRNA针对不同的菌株, 其干扰效果也具有差异性。所以RNA干扰试验需要针对目标基因合成多组SiRNA, 以便能够取得较好的干扰效果。此外一些非特异性的SiRNA也会对目标基因造成非特异性干扰, 且会随着这些SiRNA浓度的增加使干扰效果随之增加。
通过RNA干扰试验证明, SiRNA可以抑制blaKPC-2 基因的表达, 减少 blaKPC-2 mRNA的产生。由于没有进一步检测KPC酶产量的变化, 所以尚不能证明RNA干扰技术是否能直接影响细菌的耐药表型。
本研究虽然证明了SiRNA干扰可以作为一种研究细菌基因功能的分子生物学手段, 其操作性比基因敲除简单易行, 但SiRNA干扰技术运用于细菌的研究还存在许多问题, 如化学合成SiRNA的价格较贵, 管家基因和非特异性对照基因的选择也比较局限, 化学合成的SiRNA通过电转移进入细菌, 传代多次后干扰效果会消失, 针对基因不同位点的SiRNA干扰效果也具有随机性, 可参考的相关文献和试验条件也较少, 所以SiRNA运用于细菌基因的研究还需要更多的试验来完善。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|