作者简介:吴 琼,女,1977年生,博士,主管技师,主要从事细菌耐药机制研究。
探讨临床分离的对氨基糖苷类耐药的肠杆菌科细菌产16S rRNA甲基化酶状况,分析其分子流行趋势及其耐药性形成和传播的机制。
方法采用纸片扩散法筛选庆大霉素和/或阿米卡星耐药的肠杆菌科细菌;采用聚合酶链反应(PCR)扩增16S rRNA甲基化酶基因、氨基糖苷修饰酶基因、β-内酰胺酶基因;采用质粒接合试验验证16S rRNA甲基化酶的转移性;应用脉冲场凝胶电泳(PFGE)对16S rRNA甲基化酶基因阳性菌株进行分型。
结果201株对庆大霉素和/或阿米卡星耐药的肠杆菌科细菌中共检出38株16S rRNA甲基化酶阳性株(
本研究分离的肠杆菌科细菌16S rRNA甲基化酶以
To investigate the molecular epidemiological characterization and the drug resistance and prevalence mechanism of 16S rRNA methylase in aminoglycoside-resistant
Gentamicin-and or amikacin-resistant
A total of 16
氨基糖苷类抗菌药物通过与原核生物30S核糖体亚基的16S rRNA高度保守的A位点结合,干扰其蛋白质合成,导致细菌死亡而起到抗菌作用。该类药物由于抗菌作用强、抗菌谱广,对革兰阴性及阳性菌均具有良好的抗菌活性,因而广泛用于各种细菌感染的临床治疗。但随着这类药物的大量应用,细菌对氨基糖苷类药物的耐药越来越严重。产生16S rRNA甲基化酶是近年来报道的介导氨基糖苷类高水平耐药的主要机制之一[ 1, 2, 3, 4, 5, 6, 7, 8]。我们对临床分离的耐氨基糖苷类肠杆菌科细菌进行了16S rRNA甲基化酶分型,分析其分子流行趋势,以了解质粒介导的16S rRNA甲基化酶所致高水平氨基糖苷类耐药性的形成和传播机制。
一、试验菌株
选择2006年10月至2007年5月从上海交通大学医学院附属瑞金医院住院及门诊、急诊患者的各种感染标本中分离出的耐氨基糖苷类肠杆菌科细菌201株,经常规细菌鉴定及ATB半自动细菌鉴定仪确证。其中有82株分离自中段尿,35株分离自痰液,17株分离自血液,9株分离自各种导管,8株分离自胆汁,50株分离自其它标本。
质控菌株为大肠埃希菌(ATCC 25922)、肺炎克雷伯菌(ATCC 700603),购于上海市临床检验中心,大肠埃希菌 J53Azr 由复旦大学医学院附属华山医院抗生素研究所惠赠。
聚合酶链反应(polymerase chain reaction,PCR)引物由上海生物工程有限公司合成。
二、方法
1.琼脂稀释法测定最小抑菌浓度(minimum inhibitory concentration, MIC) 琼脂稀释法检测肠杆菌科细菌对8种抗菌药物的MIC值,药物包括庆大霉素、阿米卡星、哌拉西林、头孢曲松、头孢噻肟、头孢他啶、亚胺培南、环丙沙星(购自卫生部中国药品生物制品检定所)。严格按照《全国临床检验操作规程》(第3版)操作。
2.超广谱β-内酰胺酶(extended spectrum beta-lactamases,ESBLs)确证试验 纸片扩散法检测菌株对头孢他啶、头孢他啶-克拉维酸、头孢噻肟、头孢噻肟-克拉维酸的抑菌圈大小。根据美国临床实验室标准化协会(Clinical and Laboratory Standards Institute, CLSI) ESBLs判定标准,头孢他啶、头孢他啶-克拉维酸和头孢噻肟、头孢噻肟-克拉维酸中任何一项检测的结果,若加克拉维酸的抑菌圈直径比不加克拉维酸抑菌圈直径≥5 mm,则可确认为产ESBLs菌株。用肺炎克雷伯菌(ATCC 700603)作为ESBLs检测质控菌株。
3.PCR扩增16S rRNA甲基化酶基因及其他耐药基因 煮沸法提取细菌DNA。PCR引物名称、序列和扩增片段大小见表1。PCR扩增体系和过程严格按照《全国临床检验操作规程》(第3版)进行。扩增产物委托上海生物工程有限公司纯化、测序。DNA测序结果经BLAST程序(http://www.ncbi.nlm.nih.gov/BLAST/)分析,与GenBank上公布的16S rRNA甲基化酶基因及β-内酰胺酶基因进行比对。
| 表1 PCR扩增的引物名称、序列和扩增片段大小 |
4.质粒接合试验 将供体菌和受体菌大肠埃希菌J53Azr分别划线接种于血平板,35 ℃孵育过夜。次日,挑取单个菌落分别转种2 mL LB肉汤中,35 ℃振荡孵育4 h。各取0.5 mL的供体菌和受体菌加入到4 mL LB肉汤中,35 ℃静置孵育过夜,取0.1 mL的混合菌液涂布于含100 μg/mL的叠氮钠和128 μg/mL阿米卡星或1 μg/mL亚胺培南的大豆胰蛋白平板,35 ℃孵育过夜。
5.脉冲场凝胶电泳(pulsed-field gel electrophoresis,PFGE)进行同源性分析 采用XbaⅠ内切酶(Takara公司)进行全基因组酶切后,用PFGE分型[ 9, 10]。细菌划线分离于血平板,35 ℃孵育过夜。次日挑取单个菌落进行PFGE。
一、耐氨基糖苷类肠杆菌科细菌的分离情况
临床分离到的对庆大霉素和/或阿米卡星耐药的201株肠杆菌科细菌中大肠埃希菌142株、肺炎克雷伯菌35株、阴沟肠杆菌24株。其中44株对庆大霉素和阿米卡星同时耐药,包括大肠埃希菌27株、肺炎克雷伯菌8株、阴沟肠杆菌9株。
二、耐氨基糖苷类肠杆菌科细菌对15种抗菌药物的敏感性
201株耐庆大霉素和/或阿米卡星的肠杆菌科细菌对临床常用各类抗菌药物耐药较严重。3种细菌对氨苄西林的耐药率最高,为100.0%;阴沟肠杆菌和大肠埃希菌对第1、2代头孢菌素的耐药率均在98.0%以上,肺炎克雷伯菌对第1、2代头孢菌素的耐药率相对较低,为88.0%以上。阴沟肠杆菌对第3代头孢菌素的耐药率为90.0%以上,其次为大肠埃希菌(对头孢噻肟耐药率为93.0%,对头孢他啶耐药率为33.1%),最后为肺炎克雷伯菌(对头孢噻肟耐药率为82.9%,对头孢他啶耐药率为80.0%)。对于第4代头孢菌素——头孢吡肟的耐药率,阴沟肠杆菌的耐药现象较为严重,为54.2%,大肠埃希菌和肺炎克雷伯菌耐药中则相对较低,分别为31.0%和37.1%。3种细菌对酶抑制剂耐药的情况也较为普遍,尤其是阴沟肠杆菌对β-内酰胺类/酶抑制剂复合物的耐药情况最为严重,对氨苄西林-舒巴坦和哌拉西林-他唑巴坦的耐药率分别为100.0%、70.8%;其次为肺炎克雷伯菌,耐药率分别为91.4%、51.4%;大肠埃希菌耐药率分别为88.7%、5.6%。3种细菌对氨基糖苷类的耐药情况是对庆大霉素的耐药率明显高于阿米卡星:所有分离的阴沟肠杆菌对庆大霉素均耐药,对阿米卡星的耐药率为37.5%;大肠埃希菌对庆大霉素和阿米卡星的耐药率分别为97.9%、19.0%;肺炎克雷伯菌则分别为97.1%、25.7%。对复方磺胺类和氟喹诺酮类的耐药率也较高,3种细菌的耐药率分别在80.0%和65.0%以上。由统计结果可知碳青霉烯类药物是敏感率最高的药物,仅阴沟肠杆菌存在5株耐药株,耐药率为20.8%,其余2种细菌均为全部敏感。
三、ESBLs确证试验
经ESBLs确证试验,142株大肠埃希菌中有139株为产ESBLs阳性株,产酶率高达97.9%;35株肺炎克雷伯菌中有25株产ESBLs,产酶率为71.4%。
四、16S rRNA甲基化酶基因扩增及DNA测序结果
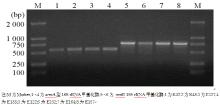
在201株对庆大霉素和/或阿米卡星耐药肠杆菌科细菌中共检出38株16S rRNA甲基化酶阳性株,其中16株(4株大肠埃希菌、6株肺炎克雷
伯菌、6株阴沟肠杆菌)为 armA基因阳性株;22株(20株大肠埃希菌、2株肺炎克雷伯菌)为 rmtB阳性株,见图1。未检测到 rmtA、 rmtC、 rmtD、 npmA基因。在其他氨基糖苷类非耐药株中均未检测到16S rRNA甲基化酶基因。PCR扩增产物经DNA双向测序证实为 armA基因和 rmtB基因,基因号分别为EU140571.1和AB263754.2。
五、β-内酰胺酶基因扩增
对38株16S rRNA甲基化酶基因阳性菌株进行β-内酰胺酶基因扩增及扩增产物双向测序,结果见表2、图2。81.6%(31/38)的 armA或 rmtB阳性菌株CTX-M型β-内酰胺酶基因呈阳性,对照菌株大肠埃希菌(ATCC 25922)PCR结果均为阴性。
| 表2 16S rRNA甲基化酶基因阳性菌株β-内酰胺酶分型 |
六、氨基糖苷修饰酶基因扩增结果
对38株16S rRNA甲基化酶基因阳性菌株进行氨基糖苷修饰酶基因扩增,结果见图3。对照菌株大肠埃希菌(ATCC 25922)PCR结果均为阴性。肺炎克雷伯菌中未检出 aac(6')- Ⅰad基因,阴沟肠杆菌中未检出 aac(6')- Ⅰad和 aac(6')- Ⅰb基因,大肠埃希菌中未检出 aac(6')- Ⅰad和 aac(6')-Ⅱ基因。 aac(3)-Ⅰ、 aac(3)-Ⅱ、 aac(6')- Ⅰb、 aac(6')-Ⅱ、 ant(3'')- Ⅰ、 ant(2'')- Ⅰ的检出率分别为26.3%、71.1%、60.5%、31.6%、76.3%和86.8%。
七、16S rRNA甲基化酶基因阳性株对8种抗菌药物的MIC值
38株16S rRNA甲基化酶基因阳性株对8种抗菌药物的耐药程度极高,见表3。其中对氨基糖苷类呈现高水平耐药,MIC50和MIC90均>256 mg/L。36株对环丙沙星耐药,耐药率高达94.7%(36/38)。5株阴沟肠杆菌对亚胺培南耐药,且该5株菌株除对多黏菌素B敏感外,对其他抗菌药物均耐药。
八、质粒接合试验结果
38株16S rRNA甲基化酶基因阳性株中有30株可在含阿米卡星和叠氮钠的筛选平板上获得接合子。其中有14株 armA阳性株,16株 rmtB阳性株。8株未能筛选到接合子。接合子抗菌药物敏感性试验显示均对庆大霉素和阿米卡星耐药,8株对环丙沙星耐药。抽提接合子质粒后,以其为模板扩增 armA基因和 rmtB基因均为阳性;扩增β-内酰胺酶基因结果显示, bla CTX-M-14、 bla TEM-1和 bla SHV-12可连同 armA或 rmtB分别转移到11、20 和7个接合子中。
| 表3 16S rRNA甲基化酶基因阳性株对8种抗菌药物的MIC值 |
九、PFGE分型结果
38株16S rRNA甲基化酶基因阳性菌株PFGE分型结果见图4。肺炎克雷伯菌、大肠埃希菌和阴沟肠杆菌分别被分为4、21和1个型别。6株阴沟肠杆菌PFGE谱型一致,可能源于同一株克隆。肺炎克雷伯菌和大肠埃希菌PFGE谱型各异,表明可能存在散在播散。
讨 论
2003年Galimand等[ 1]在分离自法国的一株肺炎克雷伯菌BM4536中发现,该菌对头孢噻肟和磺胺-甲氧苄啶耐药,对氨基糖苷类呈现异常的广泛耐药,并且该耐药表型可随质粒转移至接合试验受体菌;对链霉素-大观霉素耐药是由于 ant3''9基因介导,对其它剩余的氨基糖苷类耐药则归因于 aac3-Ⅱ和 armA基因。Liou等[ 2]研究发现, armA基因编码的ArmA蛋白使16S rRNA N7-G1405位点发生甲基化,阻碍其于4,6-脱氧链霉胺的3''氨基之间的氢键形成,并造成G1405与庆大霉素环Ⅲ之间产生空间位阻。这一甲基化将使G1405发生改变,导致氨基糖苷与译码位点不能结合,从而形成耐药。这是国际上首次报道发现的一种导致氨基糖苷类高水平耐药的质粒介导的16S rRNA甲基化酶。在后来的几年中又相继报道了5种甲基化酶基因,即 rmtA[ 3, 4]、 rmtB[ 5]、 rmtC[ 6]、 rmtD[ 7]和 npmA[ 8]。虽然在革兰阴性致病菌中新发现的这一机制在全球报道不是很多,但由于质粒介导,可在非发酵菌和肠杆菌科细菌中横向传播,也可在同一菌种间克隆传播,其危害性不容忽视。因此,本研究试图对瑞金医院分离的耐氨基糖苷类肠杆菌科细菌产质粒介导的16S rRNA甲基化酶的状况进行调查,分析其分子流行趋势、基因环境及传播机制。
本研究共收集了来自上海交通大学医学院附属瑞金医院各个临床科室分离的对庆大霉素和/或阿米卡星耐药肠杆菌科细菌201株(排除同一患者的相同菌株)。在该批肠杆菌科细菌中,共检测到38株16S rRNA甲基化酶阳性株。在44株对庆大霉素和阿米卡星同时耐药的菌株中,有6株未检测到16S rRNA甲基化酶基因,其耐药性可能由其他机制所引起。本研究对庆大霉素和/或阿米卡星非耐药株也进行了16S rRNA甲基化酶基因筛查,未检出相关基因。在所有收集的菌株中均未检测到 rmtA、 rmtC、 rmtD、 npmA基因。这表明,瑞金医院耐氨基糖苷类肠杆菌科细菌16S rRNA甲基化酶以ArmA和RmtB为流行型别。
ArmA型甲基化酶是6种16S rRNA甲基化酶中最先报道发现的。研究表明 armA基因常与CTX-M型β-内酰胺酶存在某种联系, armA基因位于接合型质粒pIP1204上,而该质粒与编码CTX-M型β-内酰胺酶的转座子相邻[ 1]。在中国台湾地区,94.3%的 armA或 rmtB阳性菌株产CTX-M型β-内酰胺酶。本研究显示,81.6%(31/38)的 armA或 rmtB阳性菌株 CTX-M型β-内酰胺酶基因扩增呈阳性。然而,同样在亚洲,一项来自韩国的调查研究发现,仅有一株16S rRNA甲基化酶阳性的肺炎克雷伯菌具有 CTX-M-14等位基因[ 11]。这一差异可能归因于地域上的不同,其真正原因有待深入研究。
rmtB最早在日本被发现[ 5]。该耐药决定因子位于非接合性大质粒上,但可经转化进入受体菌。耐药基因编码产物氨基酸序列分析显示与RmtA有82%的相似性,组氨酸标记的重组蛋白可使大肠埃希菌16S rRNA甲基化,因此将该基因命名为 rmtB,其位于Tn3结构旁。本研究中接合试验显示, rmtB基因与 armA基因相同,也可通过接合形式在不同菌株间转移,导致耐药基因的扩散。因此,在我国分离的 rmtB基因阳性菌株中,该耐药基因被证实位于可接合性质粒上。但本研究发现有8株未能得到接合子。是否该8株菌株中存在耐药决定子位于非接合性质粒上,或可通过接合形式转移至受体菌中,但不能在受体菌中复制从而丧失编码甲基化酶的功能,又或者可通过转化的形式转移到受体菌中,尚待进一步研究证实。对接合子的药物敏感试验及β-内酰胺酶等基因扩增的结果表明,β-内酰胺酶基因及氟喹诺酮类耐药决定因子可随同 armA基因和 rmtB基因在不同菌株间转移。由此可见,由质粒传播导致的对不同种类抗菌药物耐药的现象,是我们关注并采取一定措施预防和控制的重点。
质粒介导的16S rRNA甲基化酶作为一种新型氨基糖苷类耐药决定因子已引起世界的广泛关注,这提示广大医务工作者和流行病学工作者应高度重视这一耐药现象,早期筛查具有该基因的病原菌,并且采取一定措施,加强对这一类耐药菌株的监控,以防止耐药菌株广泛传播。
(收稿日期:2013-07-15)
(本文编辑:姜 敏)
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|