作者简介:罗柳林,男,1982年生,学士,主要从事临床细菌耐药机制研究。
通讯作者:应春妹,联系电话:021-68383299。
Agilent 2100分析仪在基因组外非编码重复序列片段的聚合酶链反应(REP-PCR)同源性分析中的应用并评估该系统。
方法应用REP设计引物并进行PCR扩增,用Agilent2100分析仪DL7500 Labchip芯片进行产物分析。
结果51株碳青霉烯类耐药鲍曼不动杆菌中,耐药菌株分为4大型别(A、B、C、D),其中A型共48株,分别为A1型33株,A2型13株,A3型和A4型各1株;B、C、D型各1株;18株碳青霉烯类敏感菌株中,有2株与A1型别一致,其他菌株型别都不一致。
结论基于REP-PCR的Agilent2100同源性分析具有快速、易于操作、高重复性和高分辨率、结果定量处理和多种形式输出等优点,可作为临床同源性分析鉴定和分型的一线工具。
To apply Diversilab system of the Agilent2100 bioanalyzer in repetitive-extragenome-palindrome polymerase chain reaction (REP-PCR) analysis and evaluate its performance.
MethodsPCR amplification was performed using REP as primers, its product was analyzed on DL7500 Labchip by Agilent2100 bioanalyzer.
ResultsAmong the 51 carbapenem resistance Acinetobacter baumannii, 4 epidemic patterns(A, B, C and D) were classified. 48 strains of them belonged to A pattern including sub-pattern A1(33 strains),A2(13 strains),A3(1 strain) and A4(1 strain); each one belonged to B,C and D pattern. While two belonged to the A1 pattern in 18 carbapenem sensitive isolates, others attributed to different patterns.
ConclusionsBased on the REP-PCR, Agilent2100 bioanalyzer owns many merits such as fast, feasible operating,high reproducibility and resolution,quantification of the results and many kinds of results to export. It is recommended for the first line tool in regular identification and typing research.
鲍曼不动杆菌是一种重要的医院感染致病菌, 常对多种抗菌药物耐药, 尤其是亚胺培南和美罗培南耐药株的出现, 使得临床治疗越来越困难。为阻断和控制医院感染的流行传播, 分析其基因型别及他们之间的相关性就变得尤为重要, 脉冲场凝胶电泳(PFGE)的操作流程和规则都有相应的参照, 然而其他一些非标准化检测方法会造成结果不准确、可比性不强、重复性不好等。本研究将从原核生物基因组外非编码的重复序列片段(REP)进行基因型别扩增, 在此基础上应用Agilent2100分析仪的Diversilab系统对其结果进行分析, 以此来评价该系统在同源性分析中的作用和优势。
1. 实验菌株 收集仁济医院、瑞金医院和上海市第六人民医院的耐亚胺培南和美罗培南的鲍曼不动杆菌51株, 以及仁济医院对亚胺培南和美罗培南敏感的鲍曼不动杆菌18株。
2. 抗菌药物纸片和试剂 阿米卡星、庆大霉素、氨苄西林、氨苄西林-舒巴坦、哌拉西林、头孢唑啉、头孢克罗、头孢呋辛、头孢噻肟、头孢他啶、亚胺培南、环丙沙星、复方磺胺甲口恶唑、头孢哌酮-舒巴坦、哌拉西林-他唑巴坦、头孢吡肟、美罗培南、头孢丙烯等18种抗菌药物均购于Oxoid公司; Taq酶、DNA DL2000 Maker购自Takara公司; Agilent DL7500 Marker和DNA Labchip均购于上海祥和科技有限公司。
3. 仪器 ABI 2700聚合酶链反应(PCR)扩增仪, 琼脂糖凝胶电泳仪, 天能凝胶成像分析仪, Agilent2100分析仪。
1. 抗菌药物敏感试验 纸片扩散法, 根据2005年美国临床实验室标准化研究所 (CLSI) 标准判断药敏结果。用Whonet 5.1软件进行数据处理。
2. PCR扩增 应用REP-PCR引物进行扩增, 反应体系25 μ L:5× 缓冲液5 μ L, Mg2+(3 mmol/L)3 μ L, dNTPs 混合物(各2.5 mmol/L)0.5 μ L, 引物(10 μ mol/L)0.25 μ L, 模板3 μ L, Taq酶0.3 μ L(1.5 U)。引物序列:P1为5'-IIIGCGCCGICATCAGGC-3', P2为5'-ACGTCTTATCAGGCCTAC-3'。循环条件为:94 ℃变性1 min, 40 ℃退火1 min, 65 ℃延伸8 min, 重复35个循环。
3. 同源性分析 (1)琼脂糖凝胶电泳:1.5%的琼脂糖凝胶, 100 V 1 h, 凝胶成像仪上观看结果; (2)Agilent2100的Diversilab分析: 具体操作参见“ Agilent DNA 7500 and DNA 12000 Kit Guide” 。其检测原理是:REP-PCR引物与基因组DNA在不同部位结合, 从而产生各种片段大小的扩增产物, 扩增的片段在DNA芯片装置下由微流体分开, 荧光吸收检测后Diversilab系统收集并分析数据, 同时产生相应的峰值图, 不同峰值代表不同大小DNA片段, 从而对细菌DNA进行同源性分析。
亚胺培南和美罗培南耐药的51株菌株中仅对头孢哌酮-舒巴坦有9.2%敏感, 20.8%中度敏感, 对其他17种抗菌药物全耐药。18株敏感菌株中耐药率> 80 %的有氨苄西林、头孢克罗、头孢呋辛; 耐药率在40%与80%之间的有庆大霉素、哌拉西林、头孢噻肟、头孢他啶、环丙沙星、复方磺胺甲口恶唑; < 40%的有阿米卡星、氨苄西林-舒巴坦、头孢哌酮-舒巴坦、哌拉西林-他唑巴坦、头孢吡肟; 对亚胺培南、美罗培南、头孢唑啉和头孢丙烯全敏感。
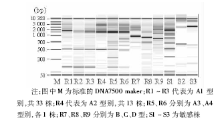
51株耐亚胺培南和美罗培南鲍曼不动杆菌可分为A、B、C、D 4个型别, 其中A型共48株(A1型33株, A2型13株, A3型和A4型各1株), B、C、D型各1株。而对亚胺培南和美罗培南敏感的18株菌株中有2株(S8、S15)与A1型别一致, 其他的型别不一致, 差异很大。7个耐药菌株型别见图1; 经过Agilent2100分析后输出的9株耐药菌株和3株敏感菌株见图2; B、C、D型耐药菌株的荧光定量吸收及相对分子质量大小图谱见图3。
多重耐药鲍曼不动杆菌的出现, 特别是对碳青霉烯类抗菌药物耐药鲍曼不动杆菌的出现, 使得临床治疗和预防感染面临极大挑战, 监测其耐药率和流行型别显得十分重要。本研究在进行同源性分析扩增的基础上, 使用了Agilent2100的Diversilab系统对其结果从凝胶图像、荧光吸收强度及其相对分子质量的大小方面进行全面的综合评价和比较。
69株鲍曼不动杆菌中, 51株耐亚胺培南和美罗培南的菌株可分为7个型别, 主要以A型别为主, 共48株; 经荧光定量吸收及相对分子质量大小图谱的比对分析发现, A2型与A1型比较在150 bp处缺失, A3型则在350和700 bp附近条带增多, A4型在850和2 000 bp附近较其增多。耐药株中的B、C、D型别在800~1 000 bp和500 bp附近条带差异较大, 且完全不同于A型。在18株敏感菌中S8和S15与A1亚型基本一致, 经分析表明在300~1 000 bp范围中A1型多了2条带的表达, 但其荧光强度依然很低, 可以考虑为一个克隆传播株。其他16株则完全不同于耐药型别, 且他们之间差异很大。
目前用于细菌同源性分析的分子生物学方法很多, 如细菌生物学分型、血清分型、噬菌体分型、多位点酶切电泳分型、核糖体分型、随机扩增片段长度多态性(RAPD)分型、REP-PCR、PFGE、扩增片段长度多态性分型(AFLP)和多位点序列分型分析(MLST)等[1, 2]。PFGE是研究最多, 也是分型分辨率最高的一种方法; AFLP则被认为是最适合于鲍曼不动杆菌分类和分型鉴定的方法[3]; 而MLST目前收集和分析的菌株主要是经过PFGE和AFLP鉴定的, 这3种方法均具有很强的可比性和高度分型能力, 尤其是MLST对于全球性流行的耐药克隆株监测具有指导作用。然而, 这些技术都有一些共同缺点, 如成本贵、技术含量高、需要专业的培训人员操作、非常耗时, 不便于操作和实行。
基于REP-PCR的Agilent2100分析仪Diversilab分析系统则是一种标准化和自动化系统, 其结果收集和分析处理不受主观限制, 40 min就可以分析12个标本; 结果具有很高的分辨率, 一般条带达到10条左右; 重复性好, 操作简单, 是一种实时定量检测[4~6]。实时是根据其片段的大小出现相应的峰, 而定量则是依照荧光强度的大小检测峰值高低, 其标准品是一种标准化操作, 且以电子文档的形式存盘, 在不同批次和不同实验室的标本进行分析比较时完全具有一样的可比性; 同时其结果既可以以凝胶图谱输出, 也可以以荧光强度为纵坐标、相对分子质量大小为横坐标的坐标图形式输出, 输出结果量化, 不同批次和时段输出也可比较。
尽管Agilent2100分析仪Diversilab系统具有很多的优点, 但其高精密度的影响因素也很多[7]。分析时只需要1 μ L的微量操作, 如偶尔的一个气泡以DNA形式加入到芯片管道后, 需要重新分析扩增产物, 这样就增加了他的检测周期和费用; Agilent2100进行分析时, 电干扰或电极上一些细小灰尘的存在也会影响结果, 这样的情况往往需要重复检测标本; 在用芯片检测的时候, 即使在标本不足的情况下, 12个加样孔和1个标准孔中必需加入标记物和凝胶荧光染料混合物, 造成试剂浪费和成本增加。
Agilent2100分析仪具有快速、易于操作、高重复性和高分辨率、结果的定量处理和多种形式输出等优点, 完全可以作为分型鉴定的一线工具, 尤其对短期内爆发流行时大量标本检测具有更大优势, 同时作为各医院常规鉴定和分型研究也具有很好的意义。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|